R(アール)は統計・データ解析・統計グラフ作成のためのオープンソースソフトです。CRAN(「シーラン」または「クラン」、Comprehensive R Archive Network)のミラーサイト(例えば https://cloud.r-project.org など)からダウンロードし、インストールしてください。Windows用、Mac用、Linux用があります。
最近では下図のような RStudio(アール・スタジオ、英語の発音により忠実に書けばアール・ステューディオまたはアール・ストゥーディオ)という統合開発環境(IDE)を介してRを使う人が増えています。Download RStudio Desktop のページからダウンロードしてインストールします。

黄色い帯の部分は参考です。読み飛ばしてもかまいません。
UNIX系OSの端末では、単に R と打ち込んで起動することもできます。カレントディレクトリがRの作業ディレクトリになります。
ESS - Emacs Speaks Statistics を使えばEmacsの中からRを使うことができます。
VS CodeにもRを使うための拡張機能があります。
Rに慣れるため、当分の間は電卓がわりにRを使ってみましょう。RまたはRStudioを起動してください。RStudioは左下の「Console」(Rコンソール)に入力します。
まず足し算です。プロンプト >
の右側に 123+456 と打ち込んでEnterキーを打ちます。[1]
の右側に出るのが答えです。
> 123+456 [1] 579
掛け算は *、割り算は /
です。計算の順序は、通常の数式と同様、掛け算・割り算が先、足し算・引き算が後になります。
> 23-4*5 [1] 3 > (23-4)*5 [1] 95 > 1/3 [1] 0.3333333 > (1/3)*3 [1] 1 > 0.3333333*3 [1] 0.9999999
C言語などでは整数の割り算は整数に切り捨てますが、Rでは / は実数(浮動小数点数)の範囲で計算します。Rでは整数の除算は %/% を使います。除算の余りは %% です。例えば 7 %/% 3 は 2 になり、7 %% 3 は 1 になります。
値は x や hoge のような変数に代入することができます。
> x = 1/3 > x*3 [1] 1
代入は x = 1/3 のような書き方のほかに x <- 1/3 や 1/3 -> x のような書き方もできます。詳しくは代入は = か <- かをご覧ください。
x = 1 / 3 のように半角スペースを入れて書いても、x=1/3 のように詰めて書いても、どちらでもかまいません。ただ、例えば x <- 1/3 を x<-1/3 のように書くと x < -1/3 と視覚的に紛らわしいので、見やすいように適宜余分なスペースを入れましょう。全角スペースは使えません。
この x のようなものを一般に「変数」と呼びますが、Rでは「オブジェクト」と呼びます。オブジェクト名は、大文字と小文字を区別します。
> x [1] 0.3333333 > X # 大文字を打ち込むと エラー: オブジェクト 'X' がありません
(英語環境では Error: object 'X' not found と表示されます。)
いげた印 # より右側はコメント(説明のための注釈)です。以下では説明のためにコメントをいろいろ書いていますが、実際に打ち込むときはコメントは必要ありません。
> pi # これは円周率 [1] 3.141593 > print(pi) # これでもいいかな [1] 3.141593 > print(pi, digits=16) # 出力する桁数を増やしたい [1] 3.141592653589793
デフォルトの表示桁数を例えば16桁にしたければ options(digits=16)
と打ち込みます。元に戻すには options(digits=7) です。
標準のRでは、浮動小数点数の精度は53ビット(ほぼ十進16桁)程度です。Rmpfr パッケージを使えば任意精度の数値が扱えます。例えば PI に1000ビット精度の円周率を代入し、それでいろいろ計算してみます:
install.packages("Rmpfr") # Rmpfrのインストール
library("Rmpfr") # このセッションでRmpfrを使う
PI = Const("pi", prec=1000) # 1000ビット精度の円周率
PI
sin(PI / 2)
任意精度の整数なら bignum パッケージの biginteger() が便利です。このパッケージには50桁精度の bigfloat() も含まれます。
Rのコンソールへの入力は、ヒストリー(履歴)機能が使えます。[↑]キー、[↓]キーで履歴を前後できます(Emacsの中では M-p、M-n)。
> pi = 3 # いたずらをしてやろう > pi # どっちのpiがアクセスできるのだろう [1] 3 > rm(pi) # piをrm(remove、削除)すると > pi # 自分の作ったpiだけ消える [1] 3.141593
pi のような既存の名前と衝突するオブジェクトを作ることは、なるべく避けたほうがいいので、新しいオブジェクトを使いたいときには、Rコンソールに打ち込んでみて「オブジェクト …… がありません」というメッセージが現れるかどうか調べるといいでしょう。
既存の1文字の名前としては c q t C D F I T があります。例えば t() は行列の転置(transpose)を求める関数です。もっとも、t = 1/3 のように数値を代入しても、関数 t() は影響を受けませんので、あまり気にする必要はありません。私も t はよく変数として使います。
pi(円周率 π = 3.14159...)は定義済みですが、e(自然対数の底 e = 2.718...)は定義されていません。
> e エラー: オブジェクト 'e' がありません
e = e1 = exp(1) ですから、指数関数 exp() を使えば e が求められます。
> exp(1) # 指数関数なら [1] 2.718282
関数の括弧を閉じないとどうなるでしょうか。
> sin(pi/2 # おっと、括弧を閉じるのを忘れてEnterを押してしまった…… +
このように、続きの入力を促すプロンプト + が出ます。その右側に、入れ忘れた ) を入力してEnterを押します。あるいは、わけがわからなくなったなら「Esc」(エスケープ)キーを押せば通常のプロンプト > に戻ります。Control+C でも止まります(Emacs中では Control-C を2回)。
こういった書き間違いの修正は面倒です。複雑な入力の際には、Rのコンソールに直接入力するのではなく、RStudioの左上のペイン(ソースエディタ)に書き、実行したい行の中にカーソルがある状態、あるいは複数行を選択した状態で、「Run」ボタン(またはCtrl+Enter、Macでは⌘+Enterでもいい)を押すのが便利です。このソースエディタに書き込んだ内容は、RStudioを終了しても保持されますが、ファイルに保存しておけば安心です。ファイル名は 何々.R のようにするのが習慣です。Rスクリプトの # から行末までは無視されますので、# を使って説明を書いておけば、後で見直すときに便利です。Emacs(ESS)の場合は、別ファイル(*.R)に書いたソースを1行ずつ実行するのは C-RET(つまりCtrl+Enter)、選択範囲を実行するのは C-c C-r、バッファ(ファイル)全体を実行するのは C-c C-b です。
RStudioでは、入力の履歴は右上ペインの「History」に残ります。「History」の中の行をダブルクリックすればコンソールに入力され、Shift+ダブルクリックすればソースエディタに入力されます。
RStudioでペイン間を移動するには、ショートカットを使うのが便利です。Ctrl+1でソースエディタ、Ctrl+2でコンソールに移動します。詳しくはRStudioの「Help」→「Keyboard Shortcuts」をご覧ください。ただ、Macでは、半角英数入力モードでも、この機能がうまく使えないことがあります。解決策としては、「環境設定」→「キーボード」→「入力ソース」の左側で「+」をクリックして「英語」→U.S. International(例えば)を追加し、「日本語」(旧「ことえり」)の入力モードの「英字」をオフにするといいでしょう。あるいは「Google日本語入力」を使うという手もあります。
ところで、答えの前にいつも出る [1] とは何でしょう?
実は、Rでは値(基本データ型)は一般にベクトル(1次元の配列)で、その要素番号(添字)が [1] から始まるのです。つまり、この数字は各行の最初の要素番号です。
> x = 5:70 # xに長いベクトル (5,6,7,8,...,70) を代入 > x # 表示させてみる [1] 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 [18] 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 [35] 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 [52] 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70
x[1] = 5,
x[2] = 6,
x[18] = 22,
x[35] = 39, ...
というわけです。
一般のベクトルを入力するときは c() という関数を使います。c
はcombineまたはconcatenate(結合する)の頭文字です。
> x = c(3.14, 2.718, 0.577) > x [1] 3.140 2.718 0.577 > x + 10 # 演算は個々の要素に作用する [1] 13.140 12.718 10.577 > x * 10 # * は掛け算の記号 [1] 31.40 27.18 5.77
x の平方根は sqrt(x) です。sq まで打って「Tab」(タブ)キーを叩くと、補完してくれます。RStudioでは簡単なヘルプも見られます。
> sqrt(x) # 関数も個々の要素に作用する [1] 1.7720045 1.6486358 0.7596052 > length(x) # ベクトルの長さ [1] 3 > sum(x) # 和 [1] 6.435 > mean(x) # 平均 [1] 2.145 > sd(x) # 標準偏差 [1] 1.374223 > x[1] # 配列の第1要素 [1] 3.14 > x[2:3] # 配列の第2〜3要素 [1] 2.718 0.577
例えば c(3,4,5,6) は 3:6
と書けます。また、c(3,5,7,9) は seq(3,9,2)
のように始値、終値、増分で指定することもできます。
c(1,1,1,1,1) は rep(1,5) と書けます。同様に、5段階のアンケート結果のように、1が2個、2が3個、…といったデータを入れるには、次のようにできます:
> x = rep(1:5, c(2,3,4,3,2)) # 1,2,3,4,5がそれぞれ2,3,4,3,2個 > x [1] 1 1 2 2 2 3 3 3 3 4 4 4 5 5 > y = rep(1:5, c(0,2,4,5,3)) > y [1] 2 2 3 3 3 3 4 4 4 4 4 5 5 5 > table(x) # 見やすい表にして表示 x 1 2 3 4 5 2 3 4 3 2 > t.test(x, y) # t検定をする
ヘルプを読むには、RStudioなら右下のペインの「Help」で検索します(現時点ではすべて英語です)。
RStudioでもRでも、次のようなコマンドでヘルプを表示できます。
help.start() … HTML版オンラインヘルプのスタートページhelp(mean) または ?mean … 特定の項目のヘルプhelp("=") や help("if") や ?"=" や ?"if" … 予約語のヘルプhelp.search("foo") または ??foo … キーワード検索apropos("foo") … 部分一致検索終了するには、[閉じる]ボタンをクリックします。または、Rのコンソールに
> q()
と打ち込むのが伝統的な方法です。q
はquit(終わる)の頭文字です。すると、標準的な設定では、
作業スペースを保存しますか?
または
Save workspace image? [y/n/c]:
と聞いてきますので、作業スペース(現在の状態)を保存して終了するなら y(はい)、保存しないで終了するなら n(いいえ)、終了をキャンセルする(終了しない)なら c(キャンセル)と答えます。通常は n(いいえ)と答えます。y(はい)と答えた場合、作業スペースの内容がRの作業ディレクトリ(working directory)の .RData というファイルに保存され、次回の起動時に自動的に読み込まれます。大量のデータを扱った場合、.RData は非常に大きくなります。y でも n でも、打ち込んだコマンドの履歴が作業ディレクトリの .Rhistory というファイルに入るはずです(入らない設定になっていることもありますので確認してください)。この .Rhistory を残しておくほうが .RData より便利です。非常に時間のかかる計算の途中でRを終了させる必要があるときは作業スペースも保存しておくほうがいいでしょう。作業ディレクトリは、メニューからも変更できますが、コマンド getwd() で調べ、setwd("...") で変更することができます。
もし履歴が自動保存されないなら、ホームディレクトリの .Rprofile というファイルに次のように書き込んでみてください:
.Last = function() { if (interactive()) try(savehistory()) }
ここで時間があれば、少しRに慣れるために、寄り道をしましょう。
RStudioなら、本格的な作業を始める際に、まず「プロジェクト」というものを作ります。これは、特定の作業ディレクトリに関連づけられるものです(「ディレクトリ」は「フォルダ」と同じ意味です)。このフォルダの中に、作業に必要なデータや作業の成果物を全部置いておくと、ファイルの管理が楽です。Rのコンソールに打ち込んだコマンドの履歴も、このディレクトリの .Rhistory というファイルに入ります。
RStudioのメニューで「File」→「New Project...」を選びます。もし「Save workspace image to ~/.RData?」などと聞いてきたなら通常は「Don't Save」と答えます。次に、新しく作業ディレクトリを作るなら「New Directory」、既存のディレクトリを使うなら「Existing Directory」を選びます。ここでは「New Directory」→「Empty Project」と選びます。すると、ディレクトリ名(Directory name)と、その親ディレクトリ(Create project as subdirectory of)を聞いてきます。親ディレクトリのデフォルトは ~(波印、チルダ)となっていますが、これはWindowsでは「ドキュメント」(C:/Users/ユーザ名/Documents)、Macでは「ホーム」(/Users/ユーザ名)という場所です。親ディレクトリのほうは通常は変更する必要はないでしょう。ディレクトリ名のほうは、プロジェクト名にもなりますので、スペースや記号を含まない英数字で簡潔な名前にするのがいいでしょう。入力したら、「Create Project」ボタンを押してください。
プロジェクトを作ると、ディレクトリ(フォルダ)が作られ、その中に プロジェクト名.Rproj というファイルと、.Rproj.user という隠しサブディレクトリができます。Rコンソールに getwd() と打ち込めば、作業ディレクトリの位置が確認できます。
ESSや、コマンド版のRを使っている場合は、自分でディレクトリを作り、その中でRを起動します。ディレクトリを移動するには setwd("...") のように打ち込みます。
データファイル類はすべてこの作業ディレクトリ以下に入れると便利です。
Rにデータを読み込ませる方法は、手入力を含め、いろいろあります。ここではまず、他ソースからのコピー&ペースト(コピペ)による方法を説明します。
以下では、コピーする元データは、テキストファイルでもWebページでもPDFでもWordでもExcelでもかまいません。必要な部分だけをコピーし、Rに貼り付ける練習をします。
使うデータは、睡眠薬の古い論文からです(Cushny, A. R. and Peebles, A. R. (1905), The action of optical isomers: II hyoscines. The Journal of Physiology 32, 501–510)。
| X | 0.7 | -1.6 | -0.2 | -1.2 | -0.1 | 3.4 | 3.7 | 0.8 | 0.0 | 2.0 |
|---|---|---|---|---|---|---|---|---|---|---|
| Y | 1.9 | 0.8 | 1.1 | 0.1 | -0.1 | 4.4 | 5.5 | 1.6 | 4.6 | 3.4 |
10人の被験者に、2種類の睡眠薬X、Yを処方して、何も飲まなかったときの睡眠時間より睡眠時間がどれだけ増えたかを示したデータです。
まず、薬Xについての10個のデータをRに読み込んで、それを x という変数に代入することにしましょう。Rのコンソールに、次のように打ち込んでください。
x = scan()
すると、
1:
というプロンプトが出ます。ここで、上の表の「X」の横の10個の数値をマウスで範囲選択し、右クリックして「コピー」します(あるいは Ctrl+C)。次に、Rコンソールを右クリックして「Paste(貼り付け)」します(あるいは Ctrl+V)。最後にEnterキーの空打ちをすると、入力が終了し、「Read 10 items」のような表示が出ます(10個の値を読み込んだ場合)。念のためRに「x」と打ち込んでEnterし、正しい値が読み込まれているか確認しましょう。
> x [1] 0.7 -1.6 -0.2 -1.2 -0.1 3.4 3.7 0.8 0.0 2.0
これでデータがRに読み込まれたことが確認できました。同じようにして、薬Yについても、y という変数に読み込んでください。
次のような命令をRコンソールにコピペしても同じことです:
x = c(0.7, -1.6, -0.2, -1.2, -0.1, 3.4, 3.7, 0.8, 0.0, 2.0) y = c(1.9, 0.8, 1.1, 0.1, -0.1, 4.4, 5.5, 1.6, 4.6, 3.4)
Rコンソールに fix(x) と打ち込むと、テキストエディタに c(0.7, -1.6, -0.2, -1.2, -0.1, 3.4, 3.7, 0.8, 0.0, 2.0) と入ったものが表示されますので、数値を編集することができます。
欠測値については NA という2文字にしておくのがデフォルトです(Not Availableの意)。
実はこのデータはデータ例としてRに含まれています。sleep とすると表示されます。解説は ?sleep で読めます。ただ、ちょっとわかりづらい並びになっているので、次のようなコマンドで x と y に分離できます:
x = with(sleep, extra[group==1]) y = with(sleep, extra[group==2])
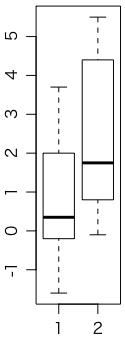
読み込んだデータを簡単なボックスプロット(箱ひげ図)で描いてみましょう:
boxplot(x, y)
そっけないグラフですね。オプションを与えれば色をつけたり項目名を入れたりできますが、とりあえずこれでいいでしょう。縦横比の調節は、[Zoom]ボタンで別窓にしてからマウスで調節します。
図ではなく、数値で最小値、25%点、中央値、平均値、75%点、最大値を求めたい場合は、次のようにします:
> summary(x) Min. 1st Qu. Median Mean 3rd Qu. Max. -1.600 -0.175 0.350 0.750 1.700 3.700
標準偏差は summary(x) で出ませんが、これは sd(x) で求められます。これは N-1 で割る方式の標準偏差です(統計の基礎参照)。
すでに統計学を勉強した人は、t検定をしてみましょう。x と y が独立な10個ずつの数だとした場合:
> t.test(y, x)
Welch Two Sample t-test
data: y and x
t = 1.8608, df = 17.776, p-value = 0.07939
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-0.2054832 3.3654832
sample estimates:
mean of x mean of y
2.33 0.75
そうではなく x と y が対応のある10個ずつの数だとした場合:
> t.test(y, x, paired=TRUE)
Paired t-test
data: y and x
t = 4.0621, df = 9, p-value = 0.002833
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
0.7001142 2.4598858
sample estimates:
mean of the differences
1.58
慣れれば次のような各群の95%信頼区間を比較する図も簡単に描けます:
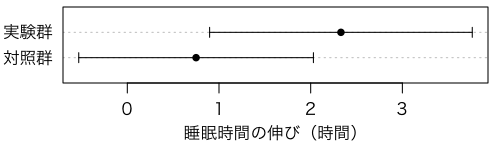
cix = t.test(x)$conf.int
ciy = t.test(y)$conf.int
dotchart(c(mean(x),mean(y)), pch=16, xlim=range(c(cix,ciy)), xlab="睡眠時間の伸び(時間)")
arrows(cix[1], 1, cix[2], 1, length=0.05, angle=90, code=3)
arrows(ciy[1], 2, ciy[2], 2, length=0.05, angle=90, code=3)
mtext(c("対照群","実験群"), side=2, at=1:2, line=0.5, las=1)
scan() を使った標準入力経由の方法は、EmacsのESS上のRでは(1行からなるデータ以外は)うまくいかないようです。その他の環境でも、scan() 以外の入力関数ではうまくいきません。このときは、クリップボード(clipboard)経由の入力を使います。何かをマウスで選択して「コピー」すると、その何かはコンピュータのメモリの「クリップボード」というところに入ります。次に別のところをクリックして「ペースト」(貼り付け)すると、クリップボードの中身がそこに入力されます。この一連の流れがコピー&ペースト(コピペ)です。
まずRのコンソールに
x = scan("clipboard")
と打ち込み、Enterキーは押さない状態で止めておきます(Macなら
x = scan(pipe("pbpaste"))
です)。その状態で、さきほどと同様にデータをコピーして、その状態で x = scan("clipboard") の最後のEnterを打ちます。すると「Read 10 items」のような表示が出て、データが読み込まれます。
Macで clipboard を指定すると、X11(X Window System)のクリップボードを読もうとします。Mac本来のクリップボード(ペーストボード、pasteboard)経由でコピー・ペーストするMacのコマンド /usr/bin/pbcopy、/usr/bin/pbpaste があります。scan(pipe("pbpaste")) はこれを(パイプ経由で)呼び出しているだけです。逆に write("hoge", pipe("pbcopy")) とすればペーストボードに「hoge」と書き出されます(コピーされます)。
atest2022.xlsx は2022年度の全国学力・学習状況調査の都道府県別正答率を収めたものです。元データは国立教育政策研究所の令和4年度 全国学力・学習状況調査 報告書・調査結果資料にあります。このデータをRに読み込んでみましょう。
まずは上述の atest2022.xlsx をダウンロード(保存)しましょう。ブラウザによりますが、デフォルトでは多分ホームディレクトリ直下の「ダウンロード」という名前のフォルダに保存されます。このままでもいいですが、プロジェクトを作成している(特定のディレクトリで仕事をしている)場合は、もしやりかたがわかるなら、データはそのプロジェクトのフォルダに保存するほうが便利です。
Excelで開いて、どのようなデータかを確認しましょう。
このExcelファイルを、RStudioを使って読み込んでみましょう。右上ペインの「Import Dataset」から「From Excel...」を選びます。
この時点で、もしライブラリreadxl、Rcppがインストールされていなかったなら、インストールの確認のメッセージが出て、インストールが始まります。
右上の「Browse...」からExcelファイルを選びます。ここではさきほど保存したatest2022.xlsxを選んでください。
同じ作業をRのコマンドで行うには、次のようにします:
library(readxl)
atest2022 = read_excel("atest2022.xlsx")
実は、ダウンロードもRから行うことができます:
download.file("https://okumuralab.org/~okumura/stat/data/atest2022.xlsx", "atest2022.xlsx")
atest2022 = read_excel("atest2022.xlsx")
これが一番簡単です。
左上ペインにデータが表示されます。
1列目は「番号」(JIS X 0401「都道府県コード」)、2列目は「都道府県」で、3列目から8列目が実際のデータです。
Excelファイルの1行目が、列名(変数名)になっています。実際のデータはExcelファイルの2行目から始まります。47都道府県あります。つまり、atest2022というデータは、47行8列の行列になっており、実際のデータ(正答率)は3〜8列目に入っています。
全体の相関図をプロットするには、Rコンソール(左下ペイン)に次のように打ち込みます:
plot(atest2022[,3:8])
[,3:8] は3〜8列目という意味です。このような指定が面倒であれば、ダウンロードしたatest2022.xlsxをExcelで編集して1列目・2列目を削除したものをRStudioで再び読み込めば、単に plot(atest2022) だけで相関図がプロットできます。
問:箱ひげ図を描いてみましょう(boxplot(atest2022[,3:8]))。科目名が全部表示されない場合は、グラフの横幅をマウスで広げてみましょう。
問:全科目の平均正答率を都道府県ごとにExcelで求め、東日本(上半分)と西日本(下半分)で統計的に有意な差があるか調べてみましょう。
問:適当な科目間で相関係数の検定をしてみましょう。
問:主成分分析や因子分析をしてみましょう。
次のようなデータがあったとしましょう。
> 名前 = c("太郎", "次郎", "花子")
> 身長 = c(168.5, 172.8, 159.0)
> 体重 = c(69.5, 75.0, 56.5)
いわゆる肥満度(BMI、body mass index)を計算してみましょう。/
は割り算、^2 は2乗を意味します。
> 体重 / (身長 / 100)^2 [1] 24.47851 25.11735 22.34880
小数点以下が長すぎるなら、round() で丸めます。
> round(体重 / (身長 / 100)^2) [1] 24 25 22 > round(体重 / (身長 / 100)^2, 1) [1] 24.5 25.1 22.3
身長・体重を一つのデータフレームにまとめると便利です。
> X = data.frame(名前, 身長, 体重) > X 名前 身長 体重 1 太郎 168.5 69.5 2 次郎 172.8 75.0 3 花子 159.0 56.5
行には 1、2、... という名前(行番号)が自動的に付きます。この部分に別の情報を入れることも可能です:
> X = data.frame(身長, 体重)
> row.names(X) = c("太郎", "次郎", "花子")
> X
身長 体重
太郎 168.5 69.5
次郎 172.8 75.0
花子 159.0 56.5
最初の二つの命令は、次のようにまとめることもできます:
> X = data.frame(身長, 体重, row.names=名前)
ただし、同じ名前の人がいると、エラーになります。データ作法として、row.names() に名前を入れることはお勧めできません。
こうしてデータフレーム X
を作れば、元の名前・身長・体重は消してもかまいません:
> rm(名前, 身長, 体重)
データフレーム X の中の 身長 を参照するには X$身長 のように書きます。
> X$身長 [1] 168.5 172.8 159.0
この X$ の部分を省略したい場合は、attach(X) と打ち込みます。逆は detach(X) です。ただし、あまりお勧めできません。
このデータフレームに性別を追加しましょう:
> X$性別 = c("M", "M", "F")
> X
名前 身長 体重 性別
1 太郎 168.5 69.5 M
2 次郎 172.8 75.0 M
3 花子 159.0 56.5 F
さらにBMIを付け加えてみましょう:
> X$BMI = round(体重 / (身長 / 100)^2) > X 名前 身長 体重 性別 BMI 1 太郎 168.5 69.5 M 24 2 次郎 172.8 75.0 M 25 3 花子 159.0 56.5 F 22
このようなデータフレームをExcelのような画面で閲覧・編集する fix()
という関数があります:
fix(X)
打ち込んでもデータ編集用の窓が現れないときは、RStudioの窓に隠れているかもしれません。Macでは、RStudioやESS上のRで fix() を使うにはX11(XQuartz)が必要です。
上のデータフレーム X は、基本的には X$名前、X$身長 などのベクトルの集まりです。ベクトルの要素はすべて同じ型を持ち、X$名前 は文字列、X$身長 は数値になるはずですが、R 4.x以前は、データフレーム化したときに X$名前 は「ファクター」(factor、カテゴリカルデータ)として格納されました。文字列を文字列のままデータフレームに格納するには、あらかじめ options(stringsAsFactors=FALSE) と打ち込んでおく必要がありました。R 4.xになってからは、これがデフォルトになりました。
名前は文字列のほうがいいのですが、性別は名義尺度のカテゴリカルデータですので、ファクターとして扱うほうがいいでしょう:
性別 = factor(c("M","M","F"))
こうすると、内部ではアルファベット順に F が1、M が2として扱われ、表示の際にそれぞれ F、M になります。後でもし数値に変換した際に男が1、女が2にしたい場合は、明示的にレベルの順序を指定します:
性別 = factor(c("M","M","F"), levels=c("M","F"))
データがある程度の量になると、Rに直接入力するより、あらかじめExcelやテキストエディタに打ち込んでからRに読み込ませるのが楽です。
例えばExcelに次のように打ち込んであるとしましょう(下の表をコピーしてWindowsのExcelにペーストしてみてください):
| 名前 | 身長 | 体重 | 性別 |
|---|---|---|---|
| 太郎 | 168.5 | 69.5 | M |
| 二郎 | 172.8 | 75.0 | M |
| 花子 | 159.0 | 56.5 | F |
ExcelからCSV(カンマ区切り)形式(*.csv)またはテキスト(タブ区切り)形式(*.txt)で保存します。ファイル名の拡張子を除いた部分はデータの名前としておくと便利です(半角英字で始まる英数字にしておくのが無難です)。ここでは data1.csv というCSV形式のファイルに保存しましょう。
あるいは、わざわざExcelを立ち上げなくても、下の4行をテキストエディタ(Windowsの「メモ帳」など)にコピペして data1.csv というファイル名で保存するのでも同じことです。
名前,身長,体重,性別 太郎,168.5,69.5,M 二郎,172.8,75,M 花子,159,56.5,F
このようにして作った data1.csv をRStudioで読み込むには、右上ペインで「Import Dataset」→「From Text File...」でファイルを選びます。
読み込むと、RStudioの左上ペインにデータのプレビューが現れます。このとき、Windows版RStudioでは日本語の部分が文字化けしますが、データとしては正しく読まれています。左下のRコンソールに data1 と打ち込んで正しく表示されることを確認してください。
Rのデフォルトの文字コードは、以前はWindowsではシフトJIS(厳密にはシフトJISをMicrosoftが拡張したCP932)でしたが、R 4.2.0からはWindowsもMacもUTF-8で統一されました(BOMなしだがBOMありも読める)。
Rを(RStudioを介さず)使っているなら、
data1 = read.csv("data1.csv")
のようにファイル名を指定すればいいのですが、data1.csv が作業ディレクトリにない状態で上のコマンドを打つとエラーになります。次のように打ち込めば、ファイルを選ぶダイアログボックスが現れるので、楽です。
data1 = read.csv(file.choose())
もちろん data1 = read.csv("D:/work/data1.csv") のようにフルパスまたは相対パスで指定すれば確実です。Windowsではパスの区切りは / または \\ とします。
文字コードが Shift JIS(CP932)の場合は data1 = read.csv("data1.csv", fileEncoding="CP932") のように文字コードを指定します。R 4.2.0 からは Windows でも "UTF-8" がデフォルトになりましたので "UTF-8"、"UTF-8-BOM" を指定する機会はなくなりました。
ファイル名のかわりにURLを指定することもできます:
data1 = read.csv("https://okumuralab.org/~okumura/stat/data/life_expectancy_Japan.csv",
comment.char="#",
fileEncoding="UTF-8")
ここでは、# で始まる行はコメントとして読み飛ばすというオプションを与えています。オンラインで提供するCSVファイルには # で始まるコメントを入れておくことがあります。
R・RStudioでは、正しく読めた場合でも、「incomplete final line」のような警告を出すことがあります。これは、最後の行に行末文字(CRまたはLFまたはCRLF)がないという意味です。正しいデータ作法としては、最後の行にも行末文字が必要ですが、Microsoft製品ではこれが付かないことがよくあります。
データフレームを読み込むには read.csv() 以外にも次の関数があります。
read.table()read.delim()RでExcelのデータを読む方法もご参照ください。
データ data1 をCSV形式のファイルに書き出すには、次のようにします(オプション row.names=FALSE で行番号の出力をしないようにしています):
write.csv(data1, "data1.csv", row.names=FALSE)
さらに、文字コードや行末を指定するには、次のようにします:
write.csv(data1, "data1.csv", row.names=FALSE, fileEncoding="CP932", eol="\r\n")
上のようにオプション fileEncoding="CP932" と eol="\r\n" を与え、ファイル名の拡張子を csv にしておけば、ダブルクリックするだけでExcelで開くことができます。一方、国際的に通用するオープンデータとするには、fileEncoding="UTF-8" のほうがいいでしょう。
保存されたファイルを見てわかるように、数値以外は "文字列" のように半角のダブルクォーテーションで囲まれます。これが目障りなら quote=FALSE オプションを与えます。
より一般には、write.table() に sep="..." のオプションを付ければ、任意の区切り文字が使えます。例えば sep="\t" とすればタブ区切りになります。
ここでは一般的な解説をします。具体的な例についてはグラフの描き方をご覧ください。
y = x2 のグラフを描いてみましょう。曲線を描く関数 curve() を使います。
curve(x^2)
x の範囲が微妙ですね。x の範囲を -2 ≦ x ≦ 2 と指定して描き直しましょう。
curve(x^2, -2, 2)
今度は y = sin(x) です。範囲は -π ≦ x ≦ π とします。
curve(sin(x), -pi, pi)
これにコサインのグラフを重ね書き(add=TRUE)しましょう。区別するため、色は青(col="blue")にします。
curve(cos(x), col="blue", add=TRUE)
x 軸、y 軸を加えます。
abline(h=0) # h は horizontal (水平) abline(v=0) # v は vertical (垂直)
タンジェントのグラフはちょっと難しいですね。明示的に x の範囲(xlim)や y の範囲(ylim)を指定するといいでしょう。
curve(tan(x), -pi/2+0.01, pi/2-0.01, xlim=c(-pi,pi), ylim=c(-2,2)) curve(tan(x), -3*pi/2+0.01, -pi/2-0.01, add=TRUE) curve(tan(x), pi/2+0.01, 3*pi/2-0.01, add=TRUE) abline(h=0, v=0) abline(v=c(-pi/2,pi/2), lty=2) # lty=2 は破線
もっと数学の教科書っぽくする方法はこちらをご覧ください。
正規分布を習った人は、正規分布の密度関数 dnorm(x) のグラフを描いてみましょう。
curve(dnorm(x), -3, 3)
少しずつ見栄えも調整してみましょう。Rのデフォルトのグラフは、私にはちょっと軸まわりが離れ過ぎのように見えます(ggplot2 の出力と比較してください)。mgp というパラメータを微調整してから描き直してみましょう:
par(mgp=c(2, 0.8, 0)) # 軸マージン(デフォルト: c(3,1,0)) curve(dnorm(x), -3, 3)
日本語でメインタイトル(main)を書き加えてみましょう。
curve(dnorm(x), -3, 3, main="正規分布")
日本語部分がトーフ(□□□□)にならないことを確認してください。
Macで、もし日本語部分がトーフになったら、
par(family="HiraginoSans-W3") # ヒラギノ角ゴシック W3 curve(dnorm(x), -3, 3, main="正規分布")
のように、フォント名を指定してください。フォント名としては、最近の Mac なら、ヒラギノ角ゴシック HiraginoSans-W0 から HiraginoSans-W9 までの10通り、ヒラギノ明朝 HiraMinProN-W3 と HiraMinProN-W6、ヒラギノ丸ゴシック HiraMaruProN-W4 などが使えます。-W の後の数字は大きいほど線が太くなります。こういった設定は、Rの初期設定ファイル ~/.Rprofile に書いておくと便利です。
上のようにすると、すべてのフォントがヒラギノ角ゴシックになってしまいます。タイトル以外を例えばHelveticaにするには、次のようにすればよいでしょう。
curve(dnorm(x), -3, 3, family="Helvetica") title(main="正規分布", family="HiraginoSans-W3", font.main=1)
title() の font.main= や、text() の font= は、1が標準、2がボールド、3がイタリック、4がボールドイタリックを表します。日本語フォントの場合は1を選ぶのが無難です(Meiryo はボールドバージョンを持っているのでデフォルトの font.main=2 のままでもかまいません)。
「正規分布」という文字の位置や大きさも直したいところですが、論文に入れる図の場合は、図の側にタイトルを入れるのではなく、本文のキャプションの側に入れるので、ここでは日本語が正しく出るかのテストだと割り切って、細かいところは気にしないことにします。
線の太さ lwd をデフォルトの1から2に変更してみます:
curve(dnorm(x), -3, 3, main="正規分布", lwd=2)
同じ座標軸にさらに曲線を書き加えるときは add=TRUE
を指定します。たとえば自由度1の t 分布の密度関数を書き加えてみます:
curve(dt(x,1), lwd=2, lty=2, col="orange", add=TRUE)
上では線の太さ lwd
以外に線のタイプ lty
と色 col
を指定しました。色は "orange"
のような色名(colors()
でリストが出ます)や、"#FFCCCC"
のような16進RGB指定ができます。モノクロ印刷の場合は gray()
関数で指定するのが便利です。この引数は 0 ≦ x ≦ 1 のグレースケールで、gray(0)
が黒、gray(1) が白です。
このほか、任意の位置に文字列を出力する text()、任意の線分を描く lines()、点を描く points()、多角形を描く polygon()
を使えば複雑な図形も描けます。
Rの図のためのウィンドウには通常「2」から始まるデバイス番号が付きます。ウィンドウをもう一つ開くには、dev.new() と打ち込みます。デバイス番号「3」のウィンドウが開き、アクティブになります。ただしRStudioのグラフィックス・デバイス(RStudioGD)は一つ(デバイス番号「2」)しか使えません。
グラフを描くコマンドの出力は、アクティブなデバイスに描かれます。例えば2番のデバイスをアクティブにするには dev.set(2) と打ち込みます。アクティブなデバイスを閉じるには dev.off() と打ち込みます(マウスで窓を閉じてもかまいません)。すべてのデバイスを閉じるには graphics.off() です。
アクティブな描画デバイスの図を例えば zu1 というオブジェクトに保存するには、zu1 = recordPlot() と打ち込みます。保存した図を再生するには replayPlot(zu1) とします。
こうして描いた図をワープロソフトなどに取り込む推奨の方法を列挙しておきます。ビットマップ図かベクトル図(Windowsならメタファイル、MacならPDF)かが選べる場合は、拡大してもぎざぎざにならないベクトル図のほうを選ぶべきです。ただし、Windowsメタファイル(WMF)は透過(transparency)が使えず、メタファイルを貼り付けたWordからのPDF出力は、よくトラブルを起こします。また、非常に要素の数の多いグラフィックでは、ベクトル図は表示に時間がかかります。
quartz() と打ち込んでQuartz窓を開けて、図を描きます。図が描けたなら、図の窓をクリックしてから ⌘+C でコピーし、他のソフトに ⌘+V で貼り付けます。QuartzはPDFをベースとしたMac標準の描画エンジンです。Rのグラフをメタファイル形式で貼り付けたWordをPDF保存すると、グラフに余分な線が入ることがあります(Bug 15307 – Extra Lines in .EMF plots with PDF Export in MS Word)。Wordのバグですので、対策としては、WordでPDF化せず、別のPDF化ツールを使います。あるいは、devEMFパッケージの emf() を使ってEMFファイルに出力し、それをWordに配置することで避けられます。
PDFなどの形式で保存するには、RStudioでは「Export」、生のRでは図のメニューから保存します。
ところが、WindowsのR・RStudioのメニューでPDF出力すると、日本語の部分がトーフになります。また、MacのRStudioでは、日本語を含んだ図をPDFで保存しようとすると、しばらく固まってしまい、エラーになります。これを防ぐには、あらかじめ
par(family="Japan1GothicBBB") # またはJapan1またはJapan1Ryumin
と打ち込んでから描画してください(警告が出ますが無視します)。フォントは埋め込まれず、"Japan1GothicBBB" ならGothicBBB-Medium、"Japan1Ryumin" ならRyumin-Light、"Japan1" ならKozMinPro-Regular-Acroの、いずれも名前参照だけになり、WindowsのAdobe Readerでは小塚、Macではヒラギノで表示されます。このようなフォントを埋め込まないPDFは、昔はファイルサイズが小さくなるのでよく使われましたが、日本語フォントがないと表示されず、今は受け取りを拒否されるのが普通になりました。
フォントを埋め込んだ印刷品質のPDFを作るには、Macでは、OSの機能(Quartz)を使ってPDFを作ることをお勧めします。Mac版Rでは標準でQuartzが使われますが、Mac版RStudioでは、上にも書いたように、あらかじめ quartz() と打ち込んでからグラフを描き、メニューからPDFで保存します。WindowsでもMacでも、後述の cairo_pdf() を使えばフォントが埋め込めます。
Rの embedFonts() 関数を使えばPDFにフォントを埋め込むことができますが、実際の作業はGhostscriptを呼び出しているだけで、Ghostscriptをインストールして、日本語フォントを正しく設定しておくことが必要です。
グラフィックの使えない端末でもできる方法です。また、自動処理にも向きます。
現時点で、Windows、Mac、Linux等で互換性があり、しかもフォントを埋め込んだPDFを生成する方法としては、おそらく cairo_pdf() が推奨できると思います。フォント名はここではWindowsで視認性の高いメイリオ("Meiryo")にしましたが、"MS Gothic" なども使えます。
cairo_pdf("hoge.pdf", width=7, height=5, family="Meiryo") # 幅・高さをインチ単位で指定
par(mgp=c(2, 0.8, 0))
curve(dnorm(x), -3, 3, main="正規分布", lwd=2)
dev.off() # ファイルを閉じる
MacではOSの機能(Quartz)を使ってコマンドでPDF出力することもできます。Macではこちらが推奨です。
quartz(type="pdf", width=7, height=5, file="hoge.pdf") par(mgp=c(2, 0.8, 0)) curve(dnorm(x), -3, 3, lwd=2, family="Helvetica") title(main="正規分布", family="HiraginoSans-W3", font.main=1) dev.off()
次のように setEPS()
と打ち込んでから postscript()
コマンドでファイル名(とフォント)を指定し、図を出力します。フォントは埋め込まれません。長い間EPSはLaTeXの推奨画像形式とされてきましたが、現在のLaTeXは最終産物がPDFの場合がほとんどですので、EPSは非推奨です。EPSを使うとLaTeXからGhostscriptを呼び出してPDFに変換するという余分な作業が入り、遅くなるだけでなく、トラブルも発生しやすくなります。
setEPS()
postscript("hoge.eps", family="Japan1GothicBBB")
curve(...) # 図を描く
dev.off() # ファイルを閉じる
印刷目的の場合は、あらかじめ ps.options()
コマンドで colormodel(色モデル)を gray(グレースケール)または cmyk(CMYKプロセスカラー)にしておくほうが安全です:
ps.options(colormodel="gray") # or "cmyk" or "rgb"
関数 png() や jpeg() が用意されています。Webで自動処理でグラフを提供するにはPNGがお薦めです。X11やcairo-develがインストールされたLinuxマシンでRをビルドした場合、cairo デバイスを使うのがいいでしょう(Macなら quartz デバイスも使えます)。
png("hoge.png", width=640, height=480, type="cairo")
...
dev.off()
LaTeX文書に挿入する図には、昔はEPSが使われましたが、今はPDF(フォントを埋め込んだもの)が推奨です。
\documentclass{jsarticle}
\usepackage[dvipdfmx]{graphicx}
\begin{document}
...
\includegraphics[width=10cm]{hoge.pdf}
...
\end{document}
これで platex で処理し、dvipdfmx でPDF化します。
余白(マージン)が大きすぎるようなら、par()
で mar パラメータを適当に設定します。下・左・上・右の順に並べます。
par(mar=c(4.2,4.1,0.4,0.5))
あるいはLaTeX側で trim
を使ってトリミングすることも可能です。こちらは左・下・右・上の順に1/72インチ単位で指定します。
\includegraphics[width=10cm,trim=125 130 150 150]{hoge.pdf}
RのtikzDeviceを使えばTikZ形式で出力することも可能です。これをLaTeX文書に挿入すれば、本文と同じフォントで統一できますし、図にTeXの数式を含めることもできます。
Rのコマンドは行末で区切られますが、;(セミコロン)で区切ることもできます。
a = 5 + 3; b = 5 - 3
は
a = 5 + 3 b = 5 - 3
と同じ意味です。
数値は 3 と書いても 3.0
と書いても倍精度実数として扱われます。整数として扱いたいなら 3L
とします。3+0i
は複素数 3 + 0i になります。値の型は typeof() 関数で調べられます:
> typeof(3) [1] "double" > typeof(3.0) [1] "double" > typeof(3L) [1] "integer" > typeof(3+0i) [1] "complex"
Rでは「3.14」のような一つの値も長さ1のベクトルです。ベクトルの型はどの要素も同じです。
> typeof(c(1,2,3)) [1] "double" > typeof(c(1,2,"a")) [1] "character" > c(1,2,"a") [1] "1" "2" "a"
演算はベクトルの要素ごとに行われます。長さが違う場合は、長い方に合わせて、短い方を何度も使います:
> 1:6 + c(10,100) [1] 11 102 13 104 15 106
Rにはたくさんのライブラリが用意されています。ライブラリの管理はマウス操作でもできますが、以下ではコマンドで行う方法を説明します。
インストールされているすべてのパッケージは library()
と打ち込むと表示されます。この中で、例えば MASS
というパッケージは“Main Package of Venables and Ripley's MASS”と説明されています。これは
Modern Applied Statistics with S-PLUS
という本(頭文字からMASSと略称する)で解説されているパッケージです。
現在読み込まれているパッケージは search() でリストされます。
> search() [1] ".GlobalEnv" "package:stats" "package:graphics" [4] "package:grDevices" "package:utils" "package:datasets" [7] "package:methods" "Autoloads" "package:base"
この中に MASS はありませんので、読み込んでみましょう。それには
library("MASS")
と打ち込みます(このダブルクォートは省略できます)。念のため search()
と打ち込むと "package:MASS" が一覧に加わります。
library()
でリストされるのはR標準のものだけです。これ以外に無数のパッケージがCRANサイトで公開されています。パッケージをインストールする前に、すでにインストールされているパッケージを念のため最新の状態に更新しておきましょう。
options(repos="https://cloud.r-project.org")
update.packages()
install.packages("パッケージ名")
こうしてから
library("パッケージ名")
と打ち込みます(このダブルクォートは省略できます)。
パッケージにはたくさんのサンプルデータが含まれているものがあります。
data()
と打ち込むと、現在読み込まれているデータ一覧が表示されます。
install.packages("パッケージ名") の逆は remove.packages("パッケージ名") です。library("パッケージ名") の逆は detach("package:パッケージ名") です。
ひとまとまりの処理をするプログラム(Rソース)を作ったなら、それを拡張子 R
のテキストファイル(例:hoge.R)に保存しておくと便利です。
hoge.R を読み込むには次の方法があります:
hoge.R をテキストエディタで開いてRコンソールにコピー&ペーストする。source("hoge.R") と打ち込む。保存場所によっては相対パス・フルパスを与える必要がある(次の「システムに依存する細かいこと」参照)。hoge.R を読み込む(環境依存)。Rソースは読み込むとすぐ実行されます。
Rソース中でデータファイルを読み込む必要があるところでは、ユーザが指定できるように、
filename = file.choose() data = read.csv(filename)
のようにすると便利です。WindowsやMacのGUIではファイルを選択するダイアログボックスが現れます。テキスト端末では「Enter file name:」と聞いてくるだけです。
Windowsではインストール時に指定した場所(例えば C:\Program Files\R あるいは C:\Users\ユーザ名\Documents\R など)の下にすべてのファイルが入ります。
Macでは /Applications/R.app と /Library/Frameworks/R.framework にインストールされます。また、自分用のライブラリの場所として ~/Library/R が使われます。起動してから作業ディレクトリなどの設定ができますし、ターミナルから
open -a R .
と打ち込めば、カレントディレクトリがRの作業ディレクトリになります。UNIX版のようにターミナル上で使うには、/Library/Frameworks/R.framework/Resources/R
のシンボリックリンクをパスの通ったところに作成しておけば、R
と打ち込むだけで起動します。
UNIX系OSで自分でコンパイルした場合、デフォルトでは /usr/local/bin と /usr/local/lib64/R、マニュアルが /usr/local/share/man/man1 に入ります。/usr/local の部分は自由に変えられますので、~/bin などに変えるのもいいでしょう。
どのシステムでも、デフォルトでは起動時に作業ディレクトリの作業スペース .RData
と履歴 .Rhistory
を読み込みます。プロジェクトごとに異なる作業ディレクトリを指定すれば、作業スペースに保存された作業内容が継続利用できます。履歴が残らない場合は設定項目を調べてください。
RStudioは、Windowsでは C:\Users\ユーザ名\AppData\Local\RStudio-Desktop に、Macでは /Users/ユーザ名/.rstudio-desktop に設定ファイルが入ります。
作業スペースに保存されたオブジェクトを一覧するには ls()
コマンドを使います(ls は list の意味です)。オブジェクトを消すには rm()
コマンドを使います(rm は remove の意味です)。
> ls() character(0) # 「何もない」という意味 > x = 3; y = 5; z = 7 > ls() [1] "x" "y" "z" > rm(x, z) > ls() [1] "y" > rm(list=ls()) # すべてを消す(注意!) > ls() character(0) # すべて消えた
消す前にオブジェクトをファイルに保存したいときは save() を使います。
> save(X, Y, file="xy.RData")
すべてのオブジェクトを保存するのは、終了のときに作業スペースを保存すると答えればいいのですが、終了せずに保存だけするには save.image()
と打ち込みます。デフォルトでは作業ディレクトリの .RData
というファイルに入りますが、ファイル名を指定することもできます:
> save.image(file="2008-12-23.RData")
Rをバッチ動作させることができます。長時間を要する計算をサーバ機のバックグラウンドで実行するのに便利です。環境変数 LANG=ja_JP.UTF8 がセットされていないと日本語がうまく扱えないかもしれません。
nohup R CMD BATCH hoge.R &
出力は hoge.Rout にされます。あるいは、そのままターミナルに出力されたほうがいいなら、Rscript hoge.R と打ち込んでも同じです(あるいは1行目に #! /usr/bin/env Rscript などと書き込んで実行許可を付けてもかまいません)。
Rのメッセージが日本語なのが嫌な人は Sys.setenv(LANGUAGE="en_US.UTF-8")
と打ち込めば英語になります。