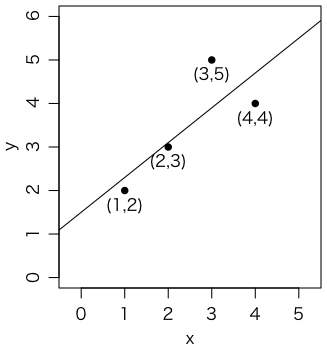

$x = (1,2,3,4)$ のとき $y = (2,3,5,4)$ になったとします(図)。このデータを例えば $y \sim ax + b$ というモデルでフィットしたいというのが回帰の問題です。ここでの「$\sim$」は「あてはめる」とか「なるべく等しくする」というつもりの記号です(詳しい意味はその都度決めます)。
念のため,右の図を描いたコード:
par(mgp=c(2,0.8,0))
plot(x, y, type="p", pch=16, xlim=c(0,5), ylim=c(0,6), asp=1)
abline(1.5, 0.8)
text(x, y, pos=1, labels=c("(1,2)","(2,3)","(3,5)","(4,4)"))
最も一般的なフィットのしかたは,残差 $y - (ax + b)$ の2乗和
\[ \sum_{i=1}^4 (y_i - (ax_i + b))^2 \]を最小にする $a$ と $b$ を求める「最小2乗法」です。
実際に上の式を最小にする $a$ と $b$ を求めてみましょう。何でも最小化してくれるRの関数 optim() を使ってみます。
x = c(1,2,3,4) # x = 1:4 とも書ける
y = c(2,3,5,4)
f = function(arg) {
a = arg[1]
b = arg[2]
t = a * x + b
return(sum((y - t)^2))
}
optim(c(1,1), f) # 初期値(a,b)=(1,1)から始めてfを最小化する
$y \sim 0.8x + 1.5$ が得られました。
実際には,わざわざこんなことをしなくても,
lm(y ~ x)
と打ち込むだけでもっと早く正確に計算してくれます。lm() という関数名はlinear modelから来ています。y ~ x は y を x の一次式でフィットするという意味です(つまり $y \sim ax + b$)。もし定数項 $b$ が不要なら lm(y ~ x - 1) とします。
ところで,一般に $x = (1,2,3,4)$ は定数と見なし,誤差は考えませんが,$y = (2,3,5,4)$ は誤差を含む測定値です。その誤差の分布は,正規分布
\[ p_i = \frac{1}{\sqrt{2\pi\sigma^2}} e^{-(y_i-\mu_i)^2/2\sigma^2} \]に従うと仮定するのが一般的です。また,誤差は互いに独立と仮定します。つまり,$y = (y_1,y_2,y_3,y_4)$ が生じる確率は積 $p_1 p_2 p_3 p_4$ に比例するとします(連続分布ですので確率というよりは確率密度ですが)。ここで,$y$ を固定して,$p_1 p_2 p_3 p_4$ をモデルパラメータ $a$,$b$ の関数と見たものを「尤度」(ゆうど)といいます。尤度が最大になるようにようにモデルのパラメータ $a$,$b$ を定めるのが最尤法(さいゆうほう)です。尤度 $p_1 p_2 p_3 p_4$ を最大にするということは,その対数 $\log(p_1 p_2 p_3 p_4)$ を最大にすることと同じことです。上の式を代入すれば,
\[ \log (p_1 p_2 p_3 p_4) = \sum_{i=1}^4 \log p_i = -\sum_{i=1}^4 \frac{(y_i-\mu_i)^2}{2\sigma^2} + \mathrm{const.} \]ですから,尤度を最大にすることは,2乗和 $\sum_{i=1}^4 (y_i-\mu_i)^2$ を最小にすることと同じです。これが,最尤法を使った最小2乗法の理屈付けです。ちなみに,より一般に分散 $\sigma^2$ も一定でない場合も含め,
\[ \sum_{i=1}^n \frac{(y_i-\mu_i)^2}{\sigma_i^2} \]の形の量を最小化するようなフィッティングを,カイ2乗最小化(chi-squared minimization,chi-square minimization)ということがあります。もしモデルが正しければ $(y_i-\mu_i)^2 / \sigma_i^2$ は標準正規分布 $N(0,1)$ の2乗になるので,それを $n$ 個加えたものは自由度 $n$ のカイ2乗分布をするはずです(実際にはモデルのパラメータ数を $n$ から引いたものが自由度です。この場合は $n-2$ が自由度です)。これを使って,あてはまりの良さを調べることができます。
モデルパラメータを $\theta$,得られたデータを $y$ とすると,$\theta$ が与えられたときに $y$ の生じる確率 $p(y \mid \theta)$ は意味が明確ですが,逆に,データ $y$ が与えられたときに $p(y \mid \theta)$ が $\theta$ の何を表すかは微妙です。ベイズ流(Bayesian,ベイジアン)の考え方では,$\theta$ にも確率分布があると考え,$\theta$ の先験的な確率分布 $p(\theta)$ と,$y$ が与えられたときの $\theta$ の確率分布 $p(\theta \mid y)$ を導入し,ベイズの定理
\[ p(\theta \mid y) = \frac{p(y \mid \theta)}{p(y)} p(\theta) \]でこれらを結びつけます。ここで $y$ を固定して $p(\theta)$ を $\theta$ によらず一定と仮定すれば,$p(y \mid \theta)$ と $p(\theta \mid y)$ は比例することになり,これをベイズ流に見た最尤法の根拠と考えることもできます。ベイジアンでは $p(\theta \mid y)$ に基づいて $\theta$ の平均値やメジアンや最頻値(モード)を求めることができますが,最尤法はこの最頻値に相当します。
上と同様に考えれば,測定値が正規分布以外の確率分布をするときでも,モデルをフィットすることができます。
ここでは,$y$ が個数を表す量である場合を考えます。例えば事故の件数や,放射線測定器が放射線をカウントする回数です。このような回数の分布は一般にポアソン分布をします。上の例で $y_i$ が平均 $\lambda_i = ax_i + b$ のポアソン分布をすると仮定すると,特定の $y_i$ の値の生じる確率は
\[ p_i = \frac{\lambda_i^{y_i} e^{-\lambda_i}}{y_i !} \]です。この特徴は,$E(y_i) = V(y_i) = \lambda_i$ つまり平均値と分散が同じ値になることです。したがって,大きい値ほど変動も大きく,単純な最小2乗法でフィットしてはいけない代表的な例です。
最尤法を使えば,さきほどと同様に
\[ \log (p_1 p_2 p_3 p_4) = \sum_{i=1}^4 (y_i \log \lambda_i - \lambda_i) + \mathrm{const.} \]を最大にすればいいことになります。さっそくやってみましょう。
f = function(arg) {
a = arg[1]
b = arg[2]
lambda = a * x + b
return(-sum(y * log(lambda) - lambda))
}
optim(c(1,1), f)
だいたい $y \sim 0.89x + 1.28$ になります。上の最小2乗法の結果とかなり違います。
実はこれも,もっと速く正確に計算する glm() という関数があります。この関数名は generalized linear model(一般化線形モデル)から来ています。
glm(y ~ x, family=poisson(link="identity"))
オプション family=poisson(link="identity") は,誤差がポアソン分布で,その平均が $\lambda_i = ax_i + b$ のように表される(左辺が $\lambda_i$ そのものである)ことを意味します。family=poisson(link="log") とすれば,$\log \lambda_i = ax_i + b$ つまり左辺は $\lambda_i$ そのものではなく対数をとったモデルになります。この対数関数(や前者の恒等関数 identity function)のように左辺に付ける関数をリンク関数といいます。なお,$\log \lambda_i = ax_i + b$ は $\lambda_i = e^{ax_i + b}$ と同じですので,$\lambda$ が負になることを心配しないでいいので,ポアソン分布のときはこちらのほうがよく使われます。単に family=poisson と書けば link="log" と見なして計算します。
ポアソン分布は $y_i$ の大きいところでは分散 $y_i \approx \lambda_i$ の正規分布で近似できます。このことを使った便法がいくつか考えられます。
ポアソン分布では,分散が期待値と等しくなります($V(y_i) = E(y_i)$)ので,ポアソン回帰の近似として,カイ2乗最小化で $\sigma_i^2 = y_i$ とすることがあります:
f = function(arg) {
a = arg[1]
b = arg[2]
lambda = a * x + b
return(sum((y - lambda)^2 / y))
}
optim(c(1,1), f)
これは次の重み付き最小2乗法と同じことです:
lm(y ~ x, weights=1/y)
結果は $y \sim 0.82x + 1.34$ になります。
「その1」の方法では,データ $y$ に0が含まれると計算できません。そこで,実現値 $y_i$ でなくモデル値 $\lambda_i = ax_i + b$ を $\sigma_i^2$ と置いて,カイ2乗最小化をすることが考えられます:
f = function(arg) {
a = arg[1]
b = arg[2]
lambda = a * x + b
return(sum((y - lambda)^2 / lambda))
}
optim(c(1,1), f)
結果は $y \sim 0.93x + 1.23$ になります。なお,この場合には lm() を使うことはできません。
r = lm(...) のように代入しておくと,回帰式の係数は r$coef に入ります。これを使って $y_i$ の予測値(ここでは $\lambda_i = ax_i + b$)を求めてもいいのですが,関数 predict(r) を使っても同じ結果が得られます。いずれにせよ,この予測値を使って,次のような繰返しをすると,何が得られるでしょうか:
w = c(1,1,1,1) # 適当な初期値
for (i in 1:10) { # 収束するまで続ける
r = lm(y ~ x, weights=w)
lambda = predict(r)
print(c(as.numeric(r$coef), -sum(y*log(lambda)-lambda)))
w = 1 / lambda
}
「その2」と同じだと思われるかもしれませんが,そうではなく,「その3」は実は本物のポアソン回帰と一致します。
どうしてかというと,こちらは $\displaystyle \sum \frac{(y_i-\lambda_i)^2}{\lambda_i}$ を分母を定数と見て最小化するので,モデルの任意のパラメータ $a$ で微分して 0 と置くときに,分子の $\lambda_i$ だけについて微分するので,最小となる点では $\displaystyle \sum \frac{2(y_i-\lambda_i)}{\lambda_i} \frac{\partial\lambda_i}{\partial a} = 0$ が成り立ちます。これは,ポアソン回帰が最大化する $\displaystyle \sum (y_i \log \lambda_i - \lambda_i)$ を $a$ で微分したものを 0 と置いた式と同じであることがすぐ確かめられます。
このループによる方法は,glm() が収束しないときにも使えますので,知っていて損はしないと思います。
このことから,nlmeパッケージの非線形な最小2乗法 gnls() でオプション weights=varPower(fixed=0.5) を与えると非線形ポアソン回帰ができる理由が推測できます。この関数は初期値によってはうまく収束しないので,なるべく正解に近い初期値を与えてやってみてください:
library(nlme)
data = data.frame(y=y, x=x)
gnls(y ~ a * x + b, data=data,
start=list(a=0.9,b=1.3),
weights=varPower(fixed=0.5))
最初のポアソン回帰の問題をもう一度,標準的な方法で,しっかり解いてみましょう。
> x = c(1,2,3,4)
> y = c(2,3,5,4)
> r = glm(y ~ x, family=poisson(link="identity"))
> summary(r)
Call:
glm(formula = y ~ x, family = poisson(link = "identity"))
Deviance Residuals:
1 2 3 4
-0.11496 -0.03194 0.51015 -0.39066
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 1.2784 1.9766 0.647 0.518
x 0.8887 0.8141 1.092 0.275
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 1.4716 on 3 degrees of freedom
Residual deviance: 0.4271 on 2 degrees of freedom
AIC: 16.779
Number of Fisher Scoring iterations: 5
ここで出てくる deviance(ディーヴィアンス,逸脱度,乖離度,尤離度)というのが,あてはまりの良さを表す量です。定義は
\[ 2 \sum ((y_i \log y_i - y_i) - (y_i \log \lambda_i - \lambda_i)) \]です。つまり,考えているモデルで最大にした対数尤度と,データとぴったり合うモデル $\lambda_i = y_i$ の対数尤度との差の2倍です。実際に計算してみましょう:
> lambda = predict(r)
> 2 * sum((y*log(y)-y) - (y*log(lambda)-lambda))
[1] 0.4270962
これが「Residual deviance」として出ているものです。これは,正規分布のときのカイ2乗統計量に相当するものです(正規分布で対数尤度に $-2$ を掛ければカイ2乗になるという話をさきほどしましたね)。これが自由度の数(データの数からモデルのパラメータの数を引いたもの)と比べて大きすぎると,あてはまりが悪いことになります。逆に,小さすぎる(0 に近すぎる)と,ポアソン分布であるというモデル自体がおかしい可能性があります。
ちなみに,$\lambda_i$ はどれも等しくてデータ $y_i$ の平均値に等しいという,まったく平らなモデル $\lambda_i = 3.5$ を考えると,
> 2 * sum((y*log(y)-y) - (y*log(3.5)-3.5))
[1] 1.471633
となります。これが「Null deviance」として出ているものです。
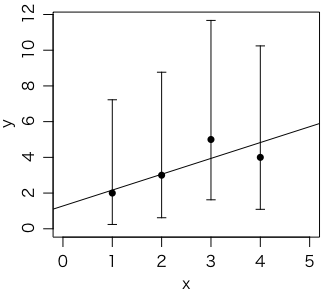
あてはまりの良さを一つの数値で表すのは便利ですが,グラフで確認するのも必要です。右の図は,データから計算した95%信頼区間と,フィットした直線を描いたものです。
これは次のようにして描きました:
l = numeric(4)
h = numeric(4)
for (i in 1:4) {
t = poisson.test(y[i])
l[i] = t$conf.int[1]
h[i] = t$conf.int[2]
}
r = glm(y ~ x, family=poisson(link="identity"))
par(mgp=c(2,0.8,0))
plot(x, y, type="p", pch=16, xlim=c(0,5), ylim=range(c(0,h)))
abline(r)
arrows(x, l, x, h, length=0.05, angle=90, code=3)
これは sapply() を使えばもうちょっとシンプルに書けます:
ci = sapply(1:4, function(i){poisson.test(y[i])$conf.int})
r = glm(y ~ x, family=poisson(link="identity"))
par(mgp=c(2,0.8,0))
plot(x, y, type="p", pch=16, xlim=c(0,5), ylim=range(c(0,ci)))
abline(r)
arrows(x, ci[1,], x, ci[2,], length=0.05, angle=90, code=3)
ちなみに,$y$ が 0 を含むことはよくありますが,$0 \times \log 0 = 0$ として計算します。具体的には
2 * sum((y*log(y)-y) - (y*log(lambda)-lambda), na.rm=TRUE)
とします。
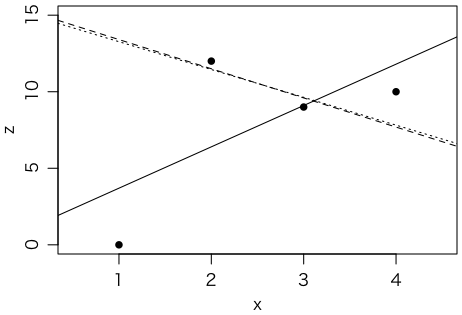
四つの町があります。各町の人口(10万人単位)は
p = c(0.01, 1, 1, 0.1)
でした。また,各町の汚染度を調べたところ,それぞれ
x = c(1, 2, 3, 4)
でした。これらの町で,ある病気で受診した患者の数が
y = c(0, 12, 9, 1)
人でした。これは10万人あたりの人数で表すと
z = y / p
です。これを $z \sim ax + b$ のような1次式でフィットしたいのですが,そのまま最小2乗法でフィットすると,図の実線のようになります。
r1 = lm(z ~ x)
plot(x, z, type="p", pch=16, xlim=c(0.5,4.5), ylim=c(0,15))
abline(r1)
でも,これでは人口1000人の村も人口10万人の都市も同じ重さでフィットされてしまいます。人口に比例した重みを付けてみましょう:
r2 = lm(z ~ x, weights=p)
abline(r2, lty=2)
破線のようになりました。
この問題をポアソン回帰で考えてみましょう。$y/p \sim ax + b$ のようにフィットしたいのですが,実際の人数は $y$ ですので,ポアソン回帰では $y \sim axp + bp$ のフィットになります。$q = xp$ と置いて glm(y ~ q + p, ...) と書けそうですが,この場合は定数項は不要なので,glm(y ~ q + p - 1, ...) という書き方をします。図は点線のようになり,ほぼ破線と一致します。
q = x * p
r3 = glm(y ~ q + p - 1, family=poisson(link="identity"))
abline(r3$coef['p'], r3$coef['q'], lty=3)
つまり,単純な最小2乗法では,汚染($x$)と発病率($z = y/p$)は正の相関をしますが,ポアソン回帰や,その近似としての重み付き最小2乗法では,負の相関になります。
ただ,この例は単なる計算のしかたを説明するためのもので,こんなに少ないデータでは,傾きの正負は有意に定まりません。実際,summary(r2) としてみると,重み付き最小2乗法の傾きの $p$ 値は 0.309 ですし,summary(r3) としてみると,ポアソン回帰の傾きのおおざっぱな $p$ 値は 0.619 です(glm(family=poisson) の $p$ 値は尤度が最大の点のまわりで正規分布近似して出しているので正確ではありません)。
[問]ポアソン分布を仮定してエラーバーを描いてみてください。
年齢調整をした場合でも,年齢調整は元の人数データの1次関数ですので,同じ方法で定式化することができます。
とりあえずスペクトルのフィット2がこの話題を扱っています。時間があればもうちょっとわかりやすいようにまとめます。
Last modified: