名古屋市のHPVVデータの解析
[おことわり] 以下はあくまでもRを使ったデータ解析法の例示で,HPVワクチンについて何らかの結論を主張するものではありません。
[2019-12-13追記] 自由記述の列229,275も含めたデータについては子宮頸がん予防接種調査の結果のPDFをCSV化をご覧ください。これ以外の列についてはすべて両者のデータが実質的に同一であることを確認しました。
データの読み込み
名古屋市のデータのCSV化で作ったCSVファイルを読み込む。
名古屋市のデータのCSV化でも書いたように,CSVに列275は含まれていない。また,列229は006001-024000の範囲がすべてNA(欠損値)になっている。これら自由記述欄以外の部分(半角数字の部分)は正しく変換されているはずである。
Nagoya-HPVV-data-01.zip を展開して kaito.csv を取り出して読む:
kaito = read.csv("kaito.csv", header=FALSE, colClasses="character", fileEncoding="UTF-8")dim(kaito)[1] 30793 274head(kaito)V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13 V14 V15 V16 V17 V18 V19 V201 000001 0 0 1 0 0 0 1 0 0 0 0 0000 0 0 0 0 0 2 00002 000002 0 1 0 0 0 0 0 0 1 0 1 0000 0 0 0 0 0 1 00003 000003 0 1 0 0 1 0 0 0 0 0 2 0000 2 0 1 0 0 1 00004 000004 0 0 1 0 0 0 0 0 0 1 1 0000 0 0 0 0 0 1 00005 000005 0 0 1 0 0 0 0 1 0 0 2 2610 1 1 0 0 0 2 26106 000006 1 0 0 0 1 0 0 0 0 0 1 0000 0 0 0 0 0 1 0000……
同じことが data.table の fread() や readr の read_csv() でもできる:
library(data.table)
kaito_fread = fread("kaito.csv", header=FALSE, colClasses="character")
library(readr)
kaito_readr = read_csv("kaito.csv", col_names=FALSE, col_types=paste0(rep("c",274), collapse=""))
readr の read_csv() では変数名が X1,X2,… となることに注意する。
子宮頸がん予防接種(HPVV)の有無を表にしてみる:
table(kaito$V233)0 1 2 3624 9245 20912 12
チェックなしが624人,なし9245人,あり20912人,両方チェックあり12人である。チェックなしや重複チェックは,他の項目の回答と照らし合わせて吟味するべきかもしれないが,ここでは単に無視する。
例:「身体が自分の意思に反して動く」
症状はいろいろある。ここではHPVV副作用としか考えられない症状(実際には思春期によく見られる)として「身体が自分の意思に反して動く」(V145)を例として調べる。なお,これはあくまでもデータ解析法の検討のための例示で,これだけから何らかの結論を導き出そうとしているわけではない。
table(kaito$V145)0 1 2259 30272 262
チェックなし259人,なし30272人,あり262人である。これをHPVV有無とクロス集計すれば
table(kaito$V233, kaito$V145)0 1 20 36 582 61 80 9107 582 143 20571 1983 0 12 0
つまり次のようになる:
| 症状なし | 症状あり | |
|---|---|---|
| HPVVなし | 9107 | 58 |
| HPVVあり | 20571 | 198 |
Fisherの正確検定をしてみる:
fisher.test(matrix(c(9107,20571,58,198),nrow=2))Fisher's Exact Test for Count Datadata: matrix(c(9107, 20571, 58, 198), nrow = 2)p-value = 0.005159alternative hypothesis: true odds ratio is not equal to 195 percent confidence interval:1.121378 2.063976sample estimates:odds ratio1.511313
非常に有意な関連がありそうである。しかし,他の変数と交絡しているかもしれないので,慎重に調べる必要がある。
まずは年齢が交絡しているかどうか調べる。まず年齢が必要である。この調査では列5〜11にそれぞれ平成6〜12年度生まれをチェックさせている。
table(rowSums(kaito[,5:11] == "1"))0 1 2456 30334 3
チェックなしが456人,二つの年度にわたってチェックしているのが3人いる。これらを除いて年齢を調べる(もっとエレガントな方法があるだろうが,面倒なので…):
birth = ifelse(rowSums(kaito[,5:11]=="1") != 1, NA,
ifelse(kaito[,5]=="1", 6,
ifelse(kaito[,6]=="1", 7,
ifelse(kaito[,7]=="1", 8,
ifelse(kaito[,8]=="1", 9,
ifelse(kaito[,9]=="1", 10,
ifelse(kaito[,10]=="1", 11,
ifelse(kaito[,11]=="1", 12, NA))))))))
よりエレガントな方法を教えていただいた:
birth <- as.numeric(apply(kaito[ ,5:11], 1, paste, collapse=""))
birth <- match(birth, 10^(6:0)) + 5
さらに別解をいただいた:
birth = as.matrix(kaito[,5:11] == "1") %*% 6:12
birth = ifelse(birth < 6 | birth > 12, NA, birth)
これは次のようにしてもよさそう。
birth = (kaito[,5:11] == "1") %*% 6:12birth = ifelse(birth %in% 6:12, birth, NA)
分布は次の通り:
table(birth, useNA="ifany")birth6 7 8 9 10 11 12 <NA>4102 4227 4235 4484 4501 4241 4544 459
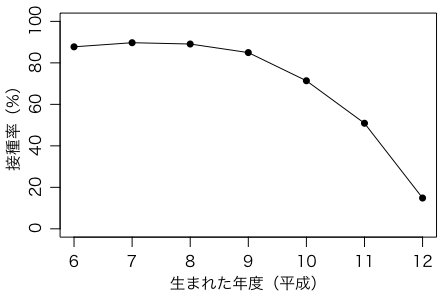
生まれた年度とHPVV接種率には強い関係がある:
s = table(kaito$V233, birth)sbirth6 7 8 9 10 11 120 56 64 90 72 96 90 1261 496 428 452 663 1260 2038 37612 3549 3735 3690 3745 3143 2112 6563 1 0 3 4 2 1 1
グラフにしてみよう:
plot(6:12, s[3,]/(s[2,]+s[3,])*100, ylim=c(0,100), type="o",
pch=16, xlab="生まれた年度(平成)", ylab="接種率(%)")
生まれた年度と「身体が自分の意思に反して動く」のクロス集計は次の通り:
u = table(kaito$V145, birth)ubirth6 7 8 9 10 11 120 22 37 26 35 48 35 331 4037 4141 4162 4414 4423 4176 44872 43 49 47 35 30 30 24
グラフにしてみよう(95%信頼区間も付けた):
plot(6:12, u[3,]/(u[2,]+u[3,])*100, ylim=c(0,2), type="o", pch=16,
xlab="生まれた年度(平成)", ylab="症状の割合(%)")
ci = sapply(1:7, function(x)binom.test(u[3,x],u[2,x]+u[3,x])$conf.int)
arrows(6:12, 100*ci[1,], 6:12, 100*ci[2,], length=0.03, angle=90, code=3)
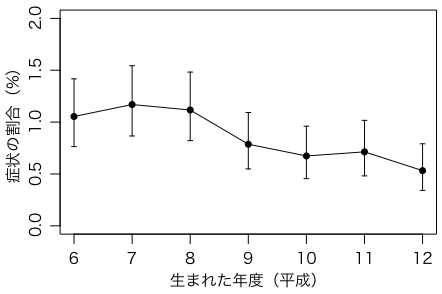
年齢が上(グラフで左側)のほうが少し多い傾向があるように見える。この傾向と,年齢が上のほうが接種率が高いこととが,年齢を交絡因子にしているのかもしれない。
横に生まれた年度(6〜12),縦に「症状なし・接種なし」「症状なし・接種あり」「症状あり・接種なし」「症状あり・接種あり」の度数を並べたクロス集計は次の通り:
t = sapply(1:7, function(x)as.numeric(table(kaito$V233, kaito$V145, birth)[2:3,2:3,x]))t[,1] [,2] [,3] [,4] [,5] [,6] [,7][1,] 486 412 447 651 1232 2014 3722[2,] 3496 3669 3627 3689 3097 2073 647[3,] 6 9 1 6 8 9 18[4,] 37 40 44 29 21 21 3
グラフにしてみよう(オレンジが接種あり,青が接種なし):
# 接種なし
plot(6:12, t[3,]/(t[1,]+t[3,])*100, ylim=c(0,2.5), type="o",
col="#0068b7", xlab="生まれた年度(平成)", ylab="症状の割合(%)")
ci = sapply(1:7, function(x)binom.test(t[3,x],t[1,x]+t[3,x])$conf.int)
arrows(6:12, 100*ci[1,], 6:12, 100*ci[2,], length=0.03, angle=90, code=3, col="#0068b7")
# 接種あり
points(6:12, t[4,]/(t[2,]+t[4,])*100, type="o", pch=16, col="#f39800")
ci = sapply(1:7, function(x)binom.test(t[4,x],t[2,x]+t[4,x])$conf.int)
arrows(6:12, 100*ci[1,], 6:12, 100*ci[2,], length=0.03, angle=90, code=3, col="#f39800")
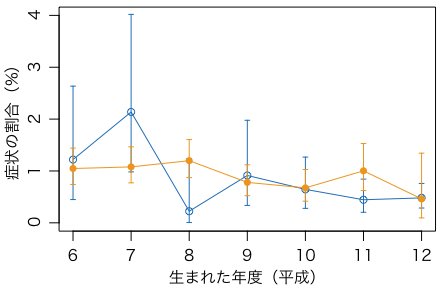
生まれた年度ごとに見ると,「身体が自分の意思に反して動く」症状が見られる割合は,接種群(オレンジ)も非接種群(青)もほぼ1%前後で生じている。両方併せれば,さきほど見たように,年齢が上(グラフで左側)のほうが少し多い傾向があるように見える。ただ,特に高年齢(左)側の非接種群(青)は人数が少ないので,変動が大きい。
メタアナリシスで解説したフォレストプロットを,生まれた年度ごとに描いてみよう:
library(metafor)
dat = escalc(measure="OR", ai=t[4,], bi=t[2,], ci=t[3,], di=t[1,])
res = rma(yi, vi, data=dat)
forest(res, slab=paste0("平成", 6:12, "年度生まれ"))
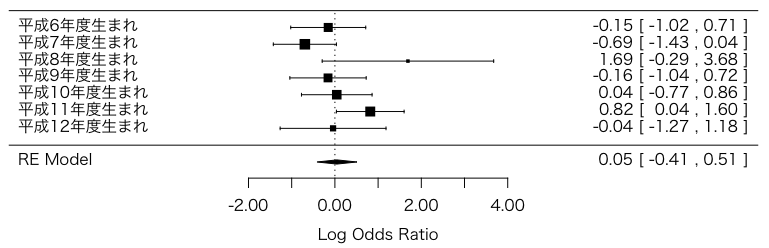
95%信頼区間を見ると,平成11年度生まれがかろうじて有意であるが,全体をまとめると対数オッズ比はほぼ 0(95%信頼区間 [-0.41, 0.51])である。
なお,ここでのフォレストプロット(および一つ前のグラフ)の目的は,全体をまとめて一つの信頼区間で表し,それで何らかの結論を出すことではなく,個々の年齢層で違いがあるのかを吟味することである。例えば,接種率が8割を超えていた平成6〜9年度生まれと,急激に減少したそれ以降とで,傾向に違いがあるのか。
例:「なかなか眠れない」
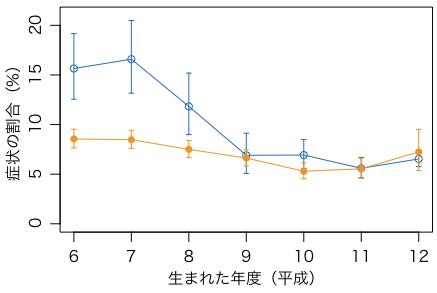
上の例は症例数が少ないのでサンプリング誤差も大きい。そこで,もうちょっと人数の多い(平凡な)症状を調べてみよう。V96(No.13)の「なかなか眠れない」である:
t = sapply(1:7, function(x)as.numeric(table(kaito$V233, kaito$V96, birth)[2:3,2:3,x]))
# 接種あり
plot(6:12, t[3,]/(t[1,]+t[3,])*100, ylim=c(0,21), type="o",
col="#0068b7", xlab="生まれた年度(平成)", ylab="症状の割合(%)")
ci = sapply(1:7, function(x)binom.test(t[3,x],t[1,x]+t[3,x])$conf.int)
arrows(6:12, 100*ci[1,], 6:12, 100*ci[2,], length=0.03, angle=90, code=3, col="#0068b7")
# 接種あり
points(6:12, t[4,]/(t[2,]+t[4,])*100, type="o", pch=16, col="#f39800")
ci = sapply(1:7, function(x)binom.test(t[4,x],t[2,x]+t[4,x])$conf.int)
arrows(6:12, 100*ci[1,], 6:12, 100*ci[2,], length=0.03, angle=90, code=3, col="#f39800")
plot(6:12, t[3,]/(t[1,]+t[3,])*100, ylim=c(0,21), type="o",
col="#0068b7", xlab="生まれた年度(平成)", ylab="症状の割合(%)")
library(metafor)
dat = escalc(measure="OR", ai=t[4,], bi=t[2,], ci=t[3,], di=t[1,])
res = rma(yi, vi, data=dat)
forest(res, slab=paste0("平成", 6:12, "年度生まれ"))
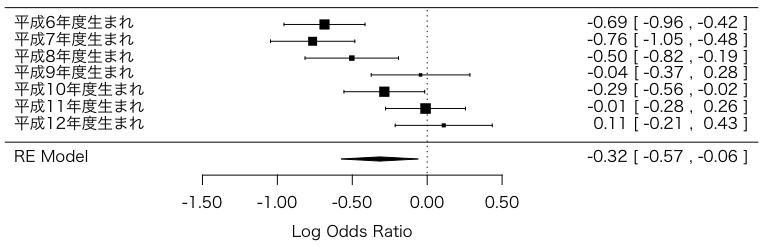
これはかなりはっきりした傾向がある。高年齢の非接種群はもともと睡眠障害があったのかもしれない(だから接種を見合わせたのかもしれない)。
(続きはそのうち書くかもしれない。)
リンク
- tonkyo_Vcさんによる解析:tonkyo_Vcさんの2016-07-03のTwilog(下のTogetterに収録された)
- Togetter: 名古屋市の子宮頸癌ワクチンアンケート調査データの解析に挑む
- かるがもクリニック: HPVワクチンのお話4:名古屋市へのコメントなど