重力波検出に成功したLIGOの SIGNAL PROCESSING WITH GW150914 OPEN DATA というチュートリアルには,Python 3を使ったデータ解析の方法が説明してある。これをRでやってみよう。
データは HDF5 形式である。RでHDF5形式を読むには,Bioconductorの rhfd5 というパッケージが便利である。まずこれをインストールしよう。
source("https://bioconductor.org/biocLite.R")biocLite("rhdf5")library("rhdf5")vignette("rhdf5")# 解説PDFを読む
CRANに最近入った h5 というライブラリも使えるかもしれない。
LIGOのチュートリアルで言及されているファイルを一つダウンロードしよう。例えば H-H1_LOSC_4_V1-1126259446-32.hdf5 というファイルは,H1側の4096HzサンプリングのGPS時刻1126259446秒から32秒間のデータである。GPS時刻とは1980-01-06 00:00:00 UTCからの秒数である(ただし閏秒を入れないので現在のUTCより17秒進んでいる)。したがってGPS時刻1126259446秒は
as.POSIXct(1126259446-17, origin="1980-01-06 00:00:00", tz="GMT")[1] "2015-09-14 09:50:29 GMT"
つまり 2015-09-14 09:50:29 UTC である。
HDF5ファイルに含まれる全データを表示したければ,次のようにする(画面が埋め尽くされるので,やらないほうがよい):
h5dump("H-H1_LOSC_4_V1-1126259446-32.hdf5")# やるなH5close()
まずはHDF5ファイルの構造を調べよう:
h5ls("H-H1_LOSC_4_V1-1126259446-32.hdf5")group name otype dclass dim0 / meta H5I_GROUP1 /meta Description H5I_DATASET STRING ( 0 )2 /meta DescriptionURL H5I_DATASET STRING ( 0 )3 /meta Detector H5I_DATASET STRING ( 0 )4 /meta Duration H5I_DATASET INTEGER ( 0 )5 /meta GPSstart H5I_DATASET INTEGER ( 0 )6 /meta Observatory H5I_DATASET STRING ( 0 )7 /meta Type H5I_DATASET STRING ( 0 )8 /meta UTCstart H5I_DATASET STRING ( 0 )9 / quality H5I_GROUP10 /quality detail H5I_GROUP11 /quality injections H5I_GROUP12 /quality/injections InjDescriptions H5I_DATASET STRING 513 /quality/injections InjShortnames H5I_DATASET STRING 514 /quality/injections Injmask H5I_DATASET INTEGER 3215 /quality simple H5I_GROUP16 /quality/simple DQDescriptions H5I_DATASET STRING 717 /quality/simple DQShortnames H5I_DATASET STRING 718 /quality/simple DQmask H5I_DATASET INTEGER 3219 / strain H5I_GROUP20 /strain Strain H5I_DATASET FLOAT 131072
最後の strain/Strain という位置にある131072個の浮動小数点数が目的のstrainデータである。次のようにしてこの部分だけ取り込む:
h1 = h5read("H-H1_LOSC_4_V1-1126259446-32.hdf5", "strain/Strain")H5close()head(h1)[1] 2.263866e-19 2.450625e-19 2.607349e-19 2.395652e-19 2.358858e-19[6] 2.275551e-19plot(h1, type="l")
別の方法として,次のようにして全データを取り込んでから,必要な部分を使うこともできる:
h1 = h5read("H-H1_LOSC_4_V1-1126259446-32.hdf5", "/")H5close()str(h1)List of 3$ meta :List of 8..$ Description : chr "Strain data time series from LIGO"..$ DescriptionURL: chr "http://losc.ligo.org/"..$ Detector : chr "H1"..$ Duration : int 32..$ GPSstart : int 1126259446..$ Observatory : chr "H"..$ Type : chr "StrainTimeSeries"..$ UTCstart : chr "2015-09-14T09:50:30"$ quality:List of 3..$ detail : NULL..$ injections:List of 3.. ..$ InjDescriptions: chr [1:5(1d)] "no cbc injections" "no burst inejctions" "no detchar injections" "no continuous wave injections" ..... ..$ InjShortnames : chr [1:5(1d)] "NO_CBC_HW_INJ" "NO_BURST_HW_INJ" "NO_DETCHAR_HW_INJ" "NO_CW_HW_INJ" ..... ..$ Injmask : int [1:32(1d)] 31 31 31 31 31 31 31 31 31 31 .....$ simple :List of 3.. ..$ DQDescriptions: chr [1:7(1d)] "data present" "passes cbc CAT1 test" "passes cbc CAT2 test" "passes cbc CAT3 test" ..... ..$ DQShortnames : chr [1:7(1d)] "DATA" "CBC_CAT1" "CBC_CAT2" "CBC_CAT3" ..... ..$ DQmask : int [1:32(1d)] 127 127 127 127 127 127 127 127 127 127 ...$ strain :List of 1..$ Strain: num [1:131072(1d)] 2.26e-19 2.45e-19 2.61e-19 2.40e-19 2.36e-19 ...head(h1$strain$Strain)[1] 2.263866e-19 2.450625e-19 2.607349e-19 2.395652e-19 2.358858e-19[6] 2.275551e-19
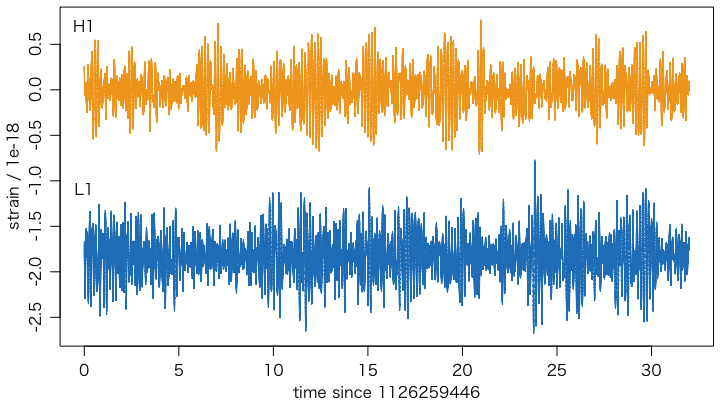
あとはL1側のデータも同様に取得し,プロットする。
t = (0:(length(h1)-1)) / 4096
H1 = h1 * 1e18
L1 = l1 * 1e18
plot(t, H1, type="l", ylim=range(c(H1,L1)), col="#f39800",
xlab="time since 1126259446", ylab="strain / 1e-18")
points(t, L1, type="l", col="#0068b7")
text(0, 0.7, "H1")
text(0, -1.1, "L1")

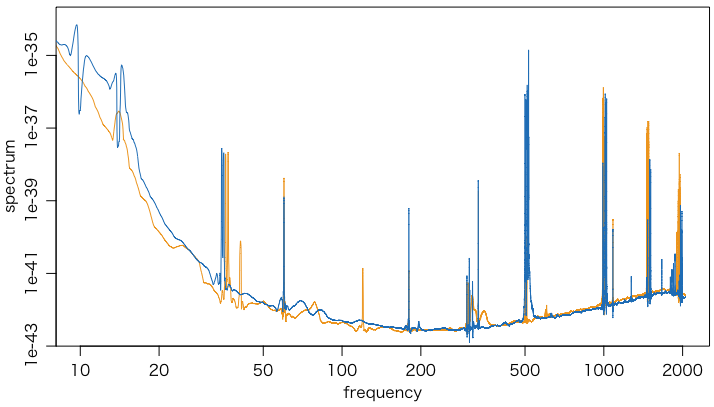
パワースペクトルを描いてみる。いろいろな方法があるが,ここでは psd という新しいパッケージを使ってみる。
install.packages("psd")
library(psd)
h1p = pspectrum(h1, x.frqsamp=4096, Nyquist.normalize=FALSE)
l1p = pspectrum(l1, x.frqsamp=4096, Nyquist.normalize=FALSE)
plot(h1p, col="#f39800")
plot(l1p, col="#0068b7", add=TRUE)
これでも十分だが,LIGOのチュートリアルに合わせて,横軸も対数目盛にしてみる:
plot(h1p$freq, h1p$spec, type="l", col="#f39800", log="xy",
xlim=c(10,2048), xlab="frequency", ylab="spectrum")
points(l1p$freq, l1p$spec, type="l", col="#0068b7")