[2018-03-21] グラフをSVG化する方法を別ページに書いた。
[2018-09-19] 全国がん罹患モニタリング集計に2018-09-15付で新しいデータが入ったので追加した。
[2018-12-28] がんに関する統計データのダウンロードに2018-11-19付で新しいデータが入ったので追加した。
[2019-06-10] 全国がん罹患モニタリング集計に新しいデータが入ったので追加した。
国立がん研究センターのがんに関する統計データのダウンロードの都道府県別死亡データから,全がん死亡数・粗死亡率・年齢調整死亡率(1995年~2017年)pref_AllCancer_mortality(1995-2017).xls,部位別75歳未満年齢調整死亡率(1995年~2017年)pref_CancerSite_mortalityASR75(1995-2017).xls をいただいてくる。
library(readxl)
par(mgp=c(2,0.8,0), las=1)
all = read_excel("pref_AllCancer_mortality(1995-2017).xls", "asr75")
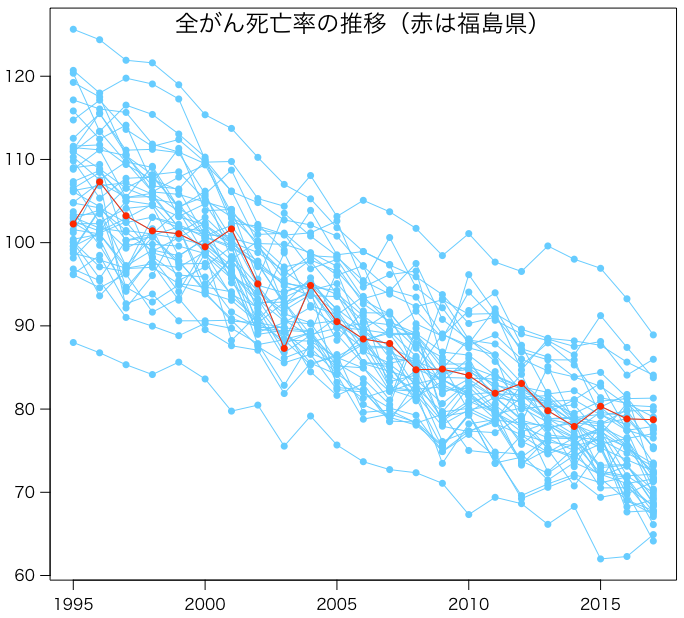
matplot(1995:2017, t(all[2:48, 4:26]), type="o", pch=16, lty=1, col="#66ccff", xlab="", ylab="")
points(1995:2017, all[8, 4:26], type="o", pch=16, col="#ff2800")
mtext("全がん死亡率の推移(赤は福島県)", line=-1.5, cex=1.5)
bui = read_excel("pref_CancerSite_mortalityASR75(1995-2017).xls", "asr75", col_types=c(rep("text",5),rep("numeric",23)))
matplot(1995:2017, t(bui[290:336, 6:28]), type="o", pch=16, lty=1, col="#66ccff", xlab="", ylab="")
points(1995:2017, bui[296, 6:28], type="o", pch=16, col="#ff2800")
mtext("胃がん死亡率の推移(赤は福島県)", line=-1.5, cex=1.5)

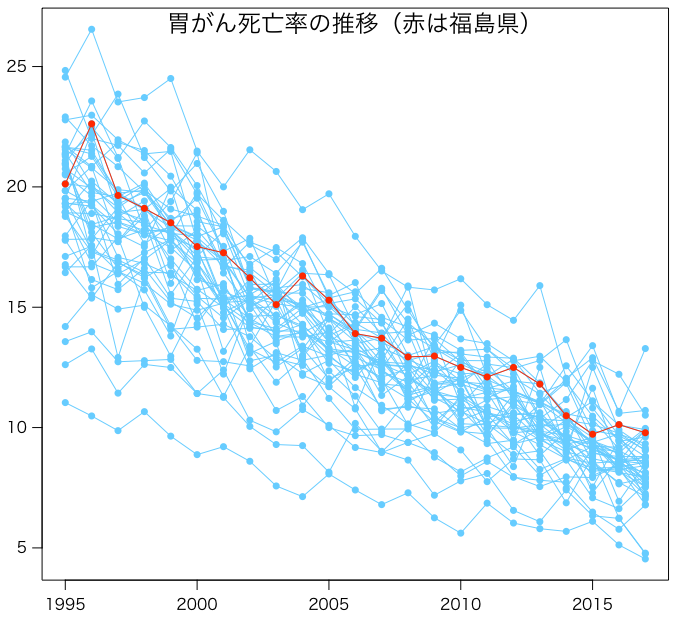
書き忘れたが,縦軸は年齢調整死亡率(男女計,人口10万対)である。
地域ごとの罹患率についてはPDFでしか公開されていないようだ。全国がん罹患モニタリング集計から,フォーマットが現在と同じ2005年以降のものをいただいてくる。ファイル名は mcij*.pdf である。これらを一括でテキストファイルにする(要 pdftotext):
for x in mcij*.pdf; do pdftotext -raw $x; done
これらのテキストから「胃 C16」という文字列の最後のものを見つけ,その後の表をコピーする。「*3」「*4」のような注が入っているものがあるので,それは削除する。できたUTF-8テキストファイルを mcij2005_C16.txt, mcij2006_C16.txt, mcij2007_C16.txt, mcij2008_C16.txt, mcij2009_C16.txt, mcij2010_C16.txt, mcij2011_C16.txt, mcij2012_C16.txt, mcij2013_C16.txt mcij2014_C16.txt mcij2015_C16.txt として置いておくので,間違いがないかPDFと比較していただければ助かる。これらをまずRで読み込む:
m05 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2005_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m06 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2006_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m07 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2007_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m08 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2008_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m09 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2009_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m10 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2010_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m11 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2011_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m12 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2012_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m13 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2013_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m14 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2014_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
m15 = read.table("https://okumuralab.org/~okumura/stat/data/mcij2015_C16.txt", fileEncoding="UTF-8", as.is=TRUE, fill=TRUE)
これらは次の項目から成る:
グラフは次のようにして描く:
x = merge(m05[,c(1,7)], m06[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m07[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m08[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m09[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m10[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m11[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m12[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m13[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m14[,c(1,7)], by="V1", all=TRUE)
x = merge(x, m15[,c(1,7)], by="V1", all=TRUE)
matplot(2005:2015, t(x[,-1]), type="o", pch=16, lty=1, col="#66ccff", xlab="", ylab="", main="男 胃 年齢調整罹患率/10万")
points(2005:2015, t(x[x[,1]=='福島県',-1]), type="o", pch=16, col="#ff2800")
y = merge(m05[,c(1,13)], m06[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m07[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m08[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m09[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m10[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m11[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m12[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m13[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m14[,c(1,13)], by="V1", all=TRUE)
y = merge(y, m15[,c(1,13)], by="V1", all=TRUE)
matplot(2005:2015, t(y[,-1]), type="o", pch=16, lty=1, col="#66ccff", xlab="", ylab="", main="女 胃 年齢調整罹患率/10万")
points(2005:2015, t(y[y[,1]=='福島県',-1]), type="o", pch=16, col="#ff2800")
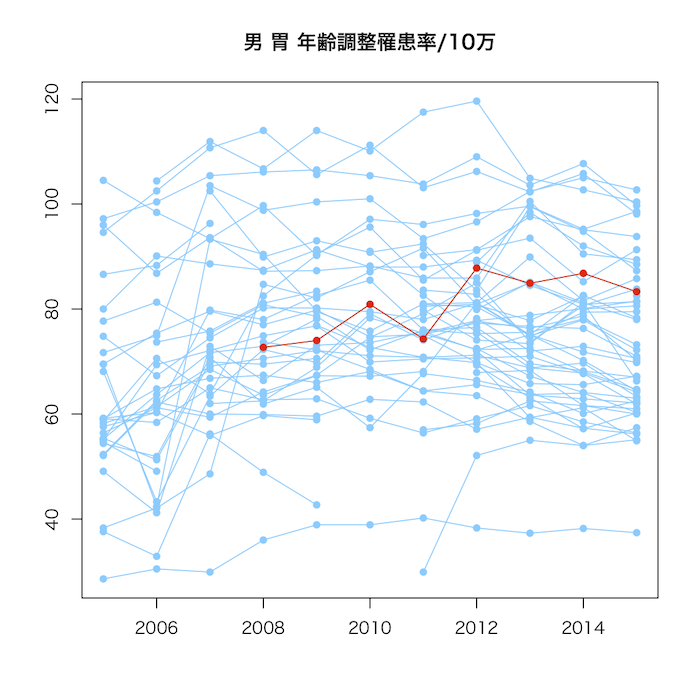
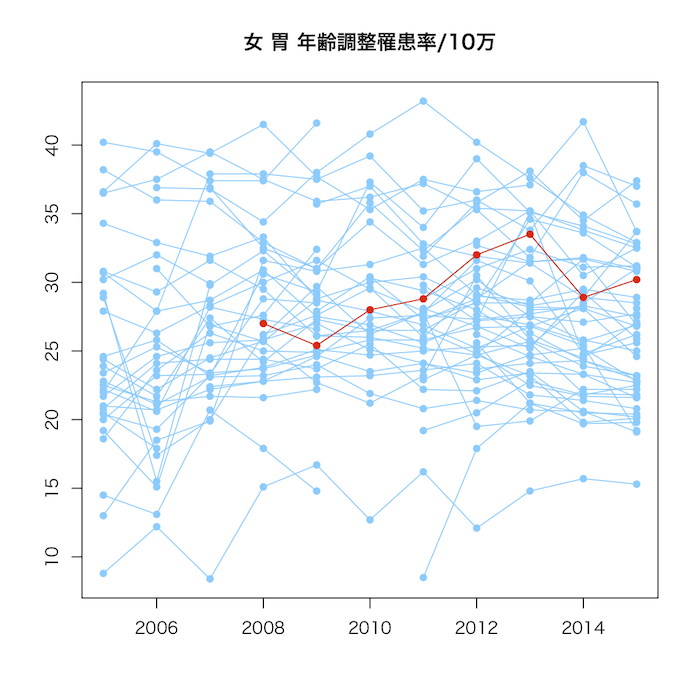
地域ごとの罹患率データは,精度がまちまちであり,地域間の比較に使うのは本来あまり望ましくない。全体の傾向を見るために使っていただきたい。
報告書 p.118 によれば,「福島県では、平成 22 年 3 月から地域がん登録事業を開始した。平成 22 年 12 月に標準データベースシステムを新規導入し、2008 年症例より集計を行っている。医療機関からの届出枚数は、年々増加し年間 25,000 枚を超えたが、その多くが拠点病院と DPC 病院からの届出となっている。地域的な偏りもあり、より高い登録精度を得るため、平成 25 年から出張採録を実施している。」とのことである。