Python でデータを読み書きするときは pandas(パンダズ)というライブラリを使うのが伝統的な方法です(今は Polars という強力な対抗馬ができました)。pandas の名前は panel data が由来だそうですが、パンダ 🐼 とかけているのでしょう。
ファイル名または URL を指定して CSV ファイルを読み込みます:
import pandas as pd
df = pd.read_csv("filename.csv")
df # 長い場合は最初と最後だけ表示してくれる
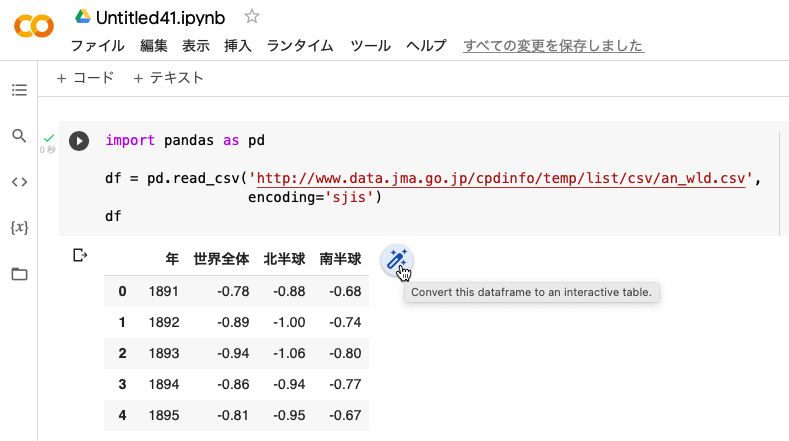
具体的に気象庁の「世界の年平均気温偏差」データを読み込んでみましょう。デフォルトの文字コードは UTF-8 ですが、これは Shift JIS ですので、オプションが必要です:
import pandas as pd
df = pd.read_csv("https://www.data.jma.go.jp/cpdinfo/temp/list/csv/an_wld.csv",
encoding="sjis")
df
Google Colaboratory なら対話型テーブルにして眺めることもできます:

読み込んだデータは DataFrame オブジェクトです:
type(df)
pandas.core.frame.DataFrame
最大表示行数・列数を調べたり変えたりしたいときは pd.options.display.max_rows、pd.options.display.max_columns を見たり代入したりします。
表示精度を変えたいときは pd.options.display.precision(デフォルト6)に桁数の目標値を代入します(IPython の %precision には影響を受けないようです)。
read_csv() でよく使うオプション:
header=None: ヘッダ行なしencoding="cp932": Shift JIS("shift_jis"、"sjis" もほぼ同じ)comment="#": # 以下をコメントと解釈するデフォルトでは次の文字列が欠測値(NaN)と判断されます:
"", "#N/A", "#N/A N/A", "#NA", "-1.#IND", "-1.#QNAN", "-NaN", "-nan", "1.#IND", "1.#QNAN", "<NA>", "N/A", "NA", "NULL", "NaN", "None", "n/a", "nan", "null"
空文字列と #N/A はExcelでも欠測値と解釈されますが、# をコメント開始文字と設定している場合(comment="#")には "#N/A" のようにダブルクォートで囲む必要があります。# を含まなければダブルクォートはあってもなくてもかまいません。
これ以外の欠測値用文字列が必要なときは na_values と keep_default_na(デフォルトは True)で指定できます。空欄だけを欠測値としたい場合は次のようにします:
df = pd.read_csv("finename.csv", na_values="", keep_default_na=False)
文字データの際には keep_default_na=False とするのが安全のようです(→ Pandasで「野根(None)」という地名が欠損値(NaN)になってしまった話)。
以下の記述について、いま確認したら、結果は 0.0 つまり誤差は出ませんでした。改善されたのでしょう。念のため残しておきます:
10進で切りの良い値なのに微妙な誤差が入ることがあります。例:
from io import StringIO data = ("A\n1258.477\n") df = pd.read_csv(StringIO(data)) df.A[0] - 1258.477 # -2.2737367544323206e-13これを避けるには
float_precision="high"またはfloat_precision="round_trip"というオプションを与えます。
pandas の read_excel() を使うのが簡単ですが、xlrd パッケージが xlsx 非対応になったため、注意が必要です。あらかじめ xlrd と openpyxl パッケージを pip install xlrd openpyxl などとしてインストールしておきます。
df = pd.read_excel("filename.xlsx") # xlsも読める
pandas によるファイル読み込みの全般については pandas の IO Tools のページをご覧ください。
xlsx の読み込みは、ほかに pylightxl という軽量なライブラリがあります。
pandas の DataFrame は R のデータフレームとほぼ同じです。簡単な解説が 10 Minutes to pandas にあります。pandasによるクエリも参考にしてください。
df # 全体を表示
df.head() # 頭の5行だけ表示
df.tail() # 末尾の5行だけ表示
df.shape # 行数・列数
df.columns # 各列の名前
df.dtypes # 各列の名前とデータ型を表示
df.index # 行の名前(デフォルトでは 0 から始まる行番号)
df.describe() # 記述統計量を表示
df.to_latex() # LaTeXのtabular形式に変換
df1 = df.dropna() # 欠測値のある行を外す
df1 = df.fillna(0) # 欠測値を0で置き換える
df1 = df.fillna(method="ffill") # 欠測値を直前の行で置き換える
df["A"] = df["A"].apply(float) # 列の各要素に関数(ここでは float())を適用
データの(0から数えて)例えば2行目から4行目を選択するには df[2:5] のようにします。データの一部の列を選択するには df.列名 あるいは df['列名'] あるいは df[['列名1','列名2']] のようにします。行列のように行名・列名で選ぶには df.loc['行名','列名']、行番号・列番号で選ぶには df.iloc[行番号,列番号] とします。複数与えるには [2,3,4] のようにリストにするか、2:5 のように範囲で表します(Pythonでは番号は0から数え、2:5 のような範囲は最後の5を含まないことに注意)。
df['列名'] はSeries(1列だけのDataFrame)という型のものです。普通のベクトル(ndarray)に直すには df['列名'].values とします。
df['列名'].value_counts(sort=False) # 列の度数分布表
df['列名'].mean() # 列の平均(median(), var(), std() なども同様)
df.groupby('列名1')['列名2'].mean() # 列名1でグループ化した列名2の平均
pd.crosstab(df['列名1'], df['列名2']) # クロス集計
場合によっては df.groupby("...").sum() のようなコードは df.groupby("...").sum(numeric_only=True) にしないと警告が出るようになりました。
この節については、いしおさんのブログDataFrameのメモリサイズを節約するが参考になります。
例えば shinjuku.csv というCSVファイルを読み込むとしましょう。
import matplotlib.pyplot as plt
import pandas as pd
import sys
url = "https://okumuralab.org/~okumura/stat/data/shinjuku.csv"
df1 = pd.read_csv(url)
df1.shape # (25705, 4)
sys.getsizeof(df1) # 2493489
df1.head() の結果は次のようになります:
datetime max min avg
0 2011/03/01 00:00 0.0370 0.0316 0.0338
1 2011/03/01 01:00 0.0368 0.0324 0.0342
2 2011/03/01 02:00 0.0357 0.0325 0.0340
3 2011/03/01 03:00 0.0371 0.0322 0.0343
4 2011/03/01 04:00 0.0363 0.0321 0.0342
まず、日付はインデックスに移動してみましょう:
df2 = pd.read_csv(url, index_col='datetime') df2.shape # (25705, 3) sys.getsizeof(df2) # 2493409
サイズの節約にはなりませんでした。df2.head() は次のようになります:
max min avg
datetime
2011/03/01 00:00 0.0370 0.0316 0.0338
2011/03/01 01:00 0.0368 0.0324 0.0342
2011/03/01 02:00 0.0357 0.0325 0.0340
2011/03/01 03:00 0.0371 0.0322 0.0343
2011/03/01 04:00 0.0363 0.0321 0.0342
日付が文字列で読み込まれてしまうのがサイズを大きくする原因です。日時データは parse_dates に指定して、pandasの64ビットの「Timestamp」型にします。この型は1970年元旦を起点としたナノ秒単位の整数で、1677年から2262年まで使えます(pd.Timestamp.min、pd.Timestamp.max)。残りの列は特に何もする必要はないのですが、ここではメモリが逼迫していると仮定して、32ビットの浮動小数点型にしてみます:
df3 = pd.read_csv(url, index_col='datetime', parse_dates=['datetime'],
dtype={'max':'float32', 'min':'float32', 'avg':'float32'})
sys.getsizeof(df3) # 514124
列指定は番号でもかまいません:
df4 = pd.read_csv(url, index_col=0, parse_dates=[0],
dtype={1:'float32', 2:'float32', 3:'float32'})
sys.getsizeof(df4) # 514124
インデックス以外の列指定が同じなら、次のようにしてもかまいません:
df5 = pd.read_csv(url, index_col=0, parse_dates=[0], dtype='float32') sys.getsizeof(df5) # 514124
データ型としては、符号付き整数 'int8'、'int16'、'int32'、'int64'、符号なし整数 'uint8'、'uint16'、'uint32'、'uint64'、浮動小数点型 'float16'、'float32'、'float64'、'float128'、論理型 'bool'(True または False)などが使えるはずです。次のような簡単なプログラムで確認できます:
from io import StringIO
data = ("A,B\n1,1.5\n2,2.5")
df = pd.read_csv(StringIO(data), dtype={0:'int8', 1:'float16'})
sys.getsizeof(df)
df.head()
データ型を指定しないと自動判断しようとしますが、うまくいかないと「Columns ... have mixed types」のような警告を出します。low_memory=False オプションで、より丁寧に自動判断してくれます。
真偽値の文字列は True、False がデフォルトですが、true_values=['Yes'], false_values=['No'] のようなオプションで指定できます。
「Timestamp」型の欠測値(空文字列、NA など)は NaN でなく NaT(Not-a-Time)になります(pd.NaT)。
日時のデータ型については日時もご参照ください。
データフレームのCSV形式での書き出しは pandas.DataFrame.to_csv() を使います。デフォルトはUTF-8ですが、Excel互換のBOM付きUTF-8(行末CRLF)でインデックスを付けずに保存するには次のようにします:
df.to_csv("filename.csv", index=False, encoding="utf_8_sig", lineterminator="\r\n")
データが10進で切りの良い値でも、微妙な誤差が入ることがあります。例えばオプション float_format="%.14g" を付けて、わざと精度を落として書き込むといった対策がありそうです。
Excel形式での書き出しは pandas.DataFrame.to_excel() です。エンジンは openpyxl または xlsxwriter が指定できます。前者のほうが多機能のようです。あらかじめ pip install openpyxl などと打ち込んでインストールしておきます。
df.to_excel("filename.xlsx")
JSON形式への変換・出力は to_json() です。lines=True で改行区切りJSON(JSONL = JSON Lines、NDJSON = Newline Delimited JSON、LDJSON = Line Delimited JSON)になります:
df = pd.DataFrame({"A":[1,2],"B":[1.5,2.5]})
df.to_json() #==> '{"A":{"0":1,"1":2},"B":{"0":1.5,"1":2.5}}'
df.to_json(orient="records")
#==> '[{"A":1,"B":1.5},{"A":2,"B":2.5}]'
df.to_json(orient="records", lines=True)
#==> '{"A":1,"B":1.5}\n{"A":2,"B":2.5}'
df.to_json('hoge.jsonl', orient="records", lines=True) # ファイルに出力
JSONL形式の読み込み:
df = pd.read_json('hoge.jsonl', orient='records', lines=True)
ちなみに、JSONファイルの操作には jq が便利です(MacならHomebrewで入れられます)。
DataFrame を dict 形式にするには:
import json
json.dumps(df.to_dict(orient="list"))
#==> '{"A": [1, 2], "B": [1.5, 2.5]}'
json.dumps(df.to_dict(orient="list"), separators=(',', ':'))
#==> '{"A":[1,2],"B":[1.5,2.5]}'
いったん dict 形式になれば、JSON ファイルへの書き出し・読み込みは次のようにできます:
with open('hoge.json', 'w', encoding='utf-8') as f:
json.dump(hoge, f)
with open('hoge.json', encoding='utf-8') as f:
hoge = json.load(f)
この open() はPython標準の関数で、文字コード encoding はシステム依存です(Python 3.11以降では locale.getencoding() で調べられます。Windows日本語環境では 'cp932'、それ以外ではだいたい 'UTF-8' です)。
全体を DataFrame に読み込まず行単位で操作するには、標準ライブラリ csv の csv.reader()、csv.writer() を使います。例:
import csv
with open('file.csv', encoding='utf-8') as f:
reader = csv.reader(f)
for row in reader:
print(row)
別の方法として、次のように少しずつ読み込む方法があります。次の例では1000行ずつ読み込んでいます。この場合 df は DataFrame を読み込むためのイテレータになります。
df = pd.read_csv('file.csv', chunksize=1000)
for chunk in df:
print(chunk.head())
Modin を使えば、import pandas as pd を import modin.pandas as pd と書き換えるだけで pandas が高速になります。
他サイトから何度も読むデータについては、キャッシュすることが推奨されます。カレントディレクトリに .web_cache というディレクトリができ、その中にキャッシュが保存されます。サイトへのアクセスは毎回しますが、前回から変わってない(ステータスコードが200ではなく304)場合はキャッシュから読みます。
import pandas as pd
from io import StringIO
import requests
from cachecontrol import CacheControl
from cachecontrol.caches.file_cache import FileCache
URL = "https://example.org/test.csv"
cache = FileCache('.web_cache', forever=True)
sess = CacheControl(requests.Session(), cache)
r = sess.get(URL)
r.encoding = "UTF-8" # または "Shift_JIS"
df = pd.read_csv(StringIO(r.text))